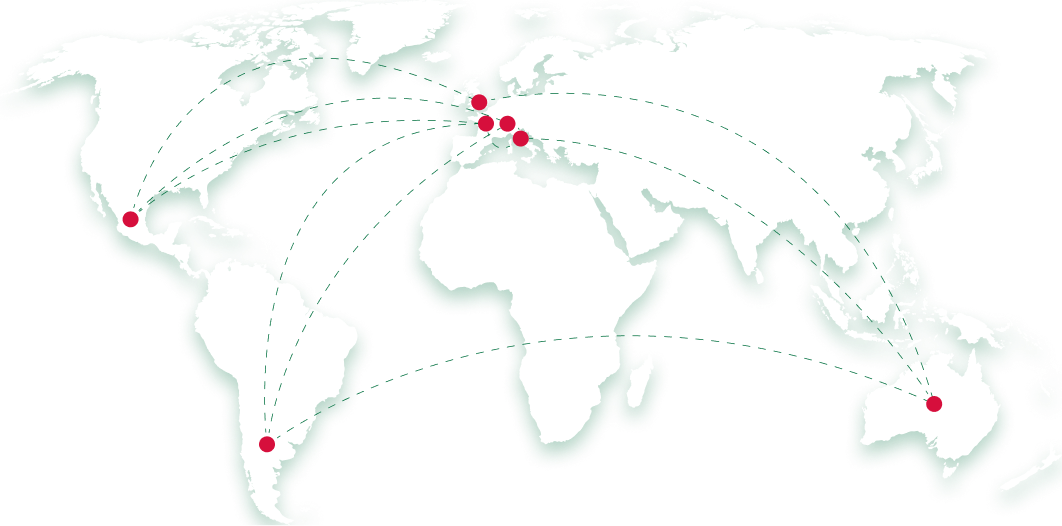
Comisionados
El término “período de misión” o “secondment period” se refiere al lapso en el que un miembro de alguna de las instituciones de la red permanece en una organización hospedadora, con el propósito de cumplir los objetivos del proyecto. Un comisionado o “secondee” es el miembro de alguna de las instituciones de la red que es albergado en la institución hospedadora.
2025
Dr. Fulvio Pupilli
IBBR, Perugia, Italia
IICAR (Rosario, Argentina), del 27 de octubre al 28 de noviembre de 2025

Dra. Viviana Echenique
CERZOS, Bahia Blanca, Argentina
IRD (Montpellier, France), del 1ro de octubre 2025 al 30 de octubre de 2025
Foto: Viviana Echenique en la UNIPG

Juan Manuel Vega, estudiante de doctorado
IICAR, Rosario, Argentina
IBBR (Perugia, Italia), del 11 de septiembre al 12 de noviembre de 2025
Juan Manuel Vega, del grupo DREP del IICAR-CONICET (Rosario, Argentina), realizará una estancia en el IBBR-CNR (Perugia, Italia) dentro del grupo del Dr. Fulvio Pupilli del 11 de septiembre al 12 de noviembre de 2025. Durante su estancia, trabajará en la mejora de un ensamblaje preliminar del genoma de Paspalum simplex mediante la realización de un andamiaje basado en el genoma de referencia de P. notatum. También contribuirá a la anotación de los genomas sexuales y apomícticos de P. simplex y a la identificación y caracterización del locus de control de la apomixis (ACL), siguiendo las estrategias desarrolladas anteriormente para P. notatum. Se llevarán a cabo análisis comparativos para evaluar la estructura del genoma en las especies de Paspalum, y se utilizarán conjuntos de datos transcriptómicos procedentes de inflorescencias para explorar la expresión génica en la región ACL. El trabajo se llevará a cabo en colaboración con Davide Perrone, Andrea Rubini y Fulvio Pupilli.
Foto: de izquirda a derecha, Fulvio Pupilli, Juan Manuel Vega & Davide Perrone

Dr. Juan Pablo A. Ortiz
IICAR, Rosario, Argentina
IRD (Montpellier, France), del 1ro de septiembre 2025 al 30 de octubre de 2025
En el marco de los WP3 y 4, Juan Pablo A. Ortiz, del IICAR-CONICET (Argentina), visitó el IRD (Montpellier, Francia) del 1 de septiembre al 30 de octubre de 2025. Durante su visita, trabajaró en el análisis de la transmisión de la aposporía (componente clave de la reproducción apomíctica) por gametos femeninos. A partir de datos de segregación generados a partir de una planta apomíctica facultativa con alta expresión de la sexualidad, se elaborará un modelo de herencia de este carácter por gametos femeninos. Los resultados obtenidos serán volcados en un manuscrito que se espera enviar a publicación a fines de año. Por otra parte, el plan de trabajo incluyó la determinación del contenido de hormonas (auxina, citoquinina, zeatina y ácido jasmónico) en los tejidos reproductivos de genotipos sexuales y apomícticos de Paspalum notatum. Los análisis bioquímicos serán realizados por Virginie Vaissayre, del IRD, utilizando la metodología de UHPLC-MS/MS. Posteriormente, el análisis de la concentración hormonal y su papel biológico en la reproducción apomíctica se realizará en colaboración con el Dr. Olivier Leblanc.
Foto: Microscopia confocal en el IRD, Lorena Siena (izquierda) y Juan Pablo Ortiz (derecha)

Dra. Maricel Podio
IICAR, Rosario, Argentina
IRD (Montpellier, France), del 1ro de septiembre 2025 al 31 de octubre de 2025
La Dra. Maricel Podio, IICAR (Zavalla, Argentina), realizó una estancia de investigación en el IRD, Montpellier, Francia, del 1 de septiembre al 31 de octubre de 2025. Durante este periodo, investigó el paisaje epigenético de Paspalum notatum mediante secuenciación de ADN genómico extraído de ovarios de genotipos tetraploides sexuales y apomícticos, utilizando la tecnología Oxford Nanopore. Para iniciar la caracterización de los epigenomas, se seleccionaron ovarios en el estadio de antesis de genotipos con diferentes niveles de expresividad de la aposporía. La secuenciación realizada durante este tiempo permitirá identificar y cuantificar las marcas de metilación del ADN (5mC y 5hmC), que luego se compararán con los perfiles de metilación del ADN de hojas para obtener información sobre los patrones epigenéticos asociados al modo de reproducción. Todos los experimentos se llevaron a cabo en colaboración dos miembros de la UMR DIADE, Julien Serret y Cédric Mariac.
Imagen: Trabajop de laboratorio en el IRD

Sol Vega, estudiante de doctorado
IICAR, Rosario, Argentina
IRD (Montpellier, France), del 1ro de septiembre 2025 al 30 de noviembre de 2025
María Sol Vega realizará una visita al IRD para profundizar la caracterización del represor de la respuesta a auxinas IAA16 en la especie modelo Arabidopsis thaliana. El ortólogo de IAA16, IAA30, está reprimido en óvulos apomícticos de Paspalum notatum en comparación con los sexuales. Las actividades de investigación incluirán la determinación del patrón de expresión espacial de IAA16 en tejido reproductivo utilizando un constructo que lleva el gen IAA16 fusionado con el gen reportero Ruby (pIAA16:IAA16:Ruby). También se obtendrán y estudiarán plantas doble homocigotas, derivadas de cruzamientos entre iaa16 y líneas marcadoras de identidad celular de gametogénesis y embriogénesis (pWOX2:CENH3, pKNU, FGR7.0, pDRN), de picos y transporte de auxina (DR5, R2D2, PIN1, aux1), y líneas mutantes para su correpresor TOPLESS y el gen asociado a la apomixis TGS1. El estudio fenotípico se llevará a cabo mediante análisis citoembriológico con microscopía DIC y análisis de líneas reporteras con microscopía de epifluorescencia y confocal. Finalmente, María Sol presentará su trabajo en la 5ª Conferencia Internacional sobre Apomixis, que se celebrará en Montpellier del 16 al 19 de septiembre. El título de su presentación es: “Auxin response repressor IAA16 defective mutants show developmental alterations in female gametophytes and embryos in Arabidopsis thaliana”.
Foto: Maria Sol Vega (izquierda) & Lorena Siena (derecha), IRD, Montpellier (France)
Dr. José Carballo
CERZOS, Bahia Blanca, Argentina
Universidad de Perugia (Italia), del 14 de agosto 2025 al 10 de octubre de 2025
Dr. Juan Pablo Selva
CERZOS, Bahia Blanca, Argentina
NIAB (Reino Unido), del 28 de julio 2025 al 10 de octubre de 2025

Dra. Lorena Siena
IICAR, Rosario, Argentina
IRD (Montpellier, Francia), del 29 de junio 2025 al 30 de noviembre de 2025
En este secondment, Lorena Siena avanzará en la caracterización funcional de los genes TRIMETHYLGUANOSINE SYNTHASE1 (TGS1) y QUI-GON JINN (QGJ) durante el desarrollo reproductivo de Arabidopsis thaliana. Las actividades se realizarán en colaboración con miembros del grupo DIADE e incluirán : analizar la expresión diferencial de genes entre plantas Col y mutantes para tgs1 en A. taliana mediante “single cell RNA sequencing “ usando la tecnologia “long reads” de ONT y ensemblar plásmidos diseñados para estudiar la función de TGS1; caracterizar la identidad celular de la megáspora funcional extra observada en óvulos de qgj usando pWOX2-CENH3-GFP como reportador; continuar con experimentos destinados a identificar la interacción potencial entre QGJ y TGS1 mediante la determinación del patrón de expresión de QGJ (pQGJ-GUS) en óvulos de tgs1, y recíprocamente, el de TGS1 (pTGS1:TGS1-GUS) en óvulos de qgj. Además, Lorena participará del Apomixis2025 International Conference (16-19 de Septiembre en Montpellier) donde fue invitada a presentar su trabajo sobre la caractérizacion de QUI-GON JINN durante el desarrollo reproductivo femenino en plantas.
Foto: Lorena, Bambouseraie de Anduze, France.

Dra. Luciana Delgado
IICAR, Rosario, Argentina
IRD (Montpellier, Francia), del 13 de junio 2025 al 10 de octubre de 2025
Durante esta última comisión de servicio dentro del WP6 del proyecto MAD, en colaboración con Daphné Autran, completaremos el análisis de datos cuantitativos de la división celular y el crecimiento celular en el primordio del óvulo de Paspalum rufum. Nuestro objetivo es analizar la dinámica del crecimiento y la división celular en contextos sexuales y apomícticos para identificar eventos diferenciales que predigan la diferenciación de células iniciales de la aposporía en las células que rodean a línea germinal sexual en una gramínea apomíctica. Además, podremos comprender cómo la arquitectura y la forma del óvulo influyen en los procesos de diferenciación celular durante la formación de la línea germinal. Durante la estancia tendremos la oportunidad de presentar y discutir nuestros resultados recientes en la “V International Conference on Apomixis”, que se realizará en septiembre de 2025 en Montpellier, dando cierre a este fructífero proyecto.
Imagen: Luciana frente a los invernaderos del IRD

Dr. Michele Bellucci
IBBR, Perugia, Italia
IICAR (Rosario, Argentina), del 29 de abril 2025 al 30 de mayo de 2025
El 29 de abril de 2025, el Dr. Michele Bellucci del CNR-IBBR, Perugia, Italia, comenzó una comisión de servicio en el IICAR hasta el 30 de mayo de 2025. Trabajará en la transformación de Paspalum notatum mediante bombardeo de partículas, con el fin de inhibir la expresión del gen PnIAA30 en plantas tetraploides sexuales y apomícticas mediante ARNi. El vector de inhibición se ensambló en el CNR-IBBR y contiene una región del gen PnIAA30 clonada en la orientación opuesta y el gen bar como marcador seleccionable. Nuestras observaciones mostraron que la expresión de PnIAA30 está reducida en los ovocitos apomícticos en comparación con los ovocitos sexuales. Nuestra hipótesis es que la inhibición de PnIAA30 (así como de algunos de sus paralogos) podría desencadenar el desarrollo de embriones partenogenéticos en plantas sexuales y/o aumentar la tasa de partenogénesis en genotipos apomícticos. Todos los experimentos se llevarán a cabo con la Dra. Carolina M. Colono y el Dr. Juan Pablo A. Ortiz.
Imagen: Carolina Colono, IICAR (izquierda) & Michele Bellucci, IBBR (derecha)
Dr. Fulvio Pupilli
IBBR, Perugia, Italia
IICAR (Rosario, Argentina), del 5 de abril de 2025 al 28 de marzo de 2025

Dr. Alessandro Camperio
Universidad de Milano, Italia
CERZOS (Bahia Blanca, Argentina), del 17 de marzo 2025 al 15 de abril de 2025
Durante esta comisión de servicio, recibí formación en la realización de análisis bioinformáticos sobre un conjunto de datos de secuenciación de ARN de núcleo único generados a partir de ovarios de Oryza sativa (muestreados en diferentes etapas de desarrollo) bajo la supervisión del Dr. José Carballo. El objetivo era adquirir conocimientos sobre el análisis del transcriptoma de un solo núcleo y estudiar la expresión de genes ARF seleccionados, identificados en otras especies como genes putativos implicados en la aposporia y la diplosporia. Como parte de este trabajo, traté de generar perfiles transcriptómicos de alta resolución que captaran la dinámica de expresión génica de las poblaciones de células gametofíticas y esporofíticas en rápida evolución durante el desarrollo del óvulo. Estos datos transcriptómicos de núcleo único se utilizarán con el objetivo más amplio de compararlos con otros conjuntos de datos de especies de Paspalum y Eragrostis. Este marco comparativo se diseñó para comprender mejor el panorama molecular descubierto en el arroz, con un enfoque particular en la detección de genes expresados en el nucelo y tejidos relacionados.
Imagen (de izquierda a derecha): Viviana Echenique, Alessandro Mariani y Alessandro Camperio.
Granpierro Marconi, MSc
Universidad de Perugia, Italia
Universidad de Adelaide (Australia), del 5 de enero de 2025 al 21 de marzo de 2025
2024
Dr. Alice Casati
Universidad de Milano, Italia
Unidad de Genómica Avanzada (Cinvestav, Mexico), del 25 de noviembre 2024 al 15 de febrero 2025

Dr. Alessandro Mariani
Universidad de Perugia, Italia
CERZOS (Bahia Blanca, Argentina), del 13 de noviembre 2024 al 26 de mayo 2025
Durante mi estancia en CERZOS-CONICET en Bahía Blanca, contribuí al proyecto Mechanisms of Apomictic Development (MAD) investigando la regulación genética y epigenética de la apomixis en las plantas. Mi investigación se centró en mapear los loci asociados con los rasgos apomícticos mediante el análisis de QTL en poblaciones segregadas, identificando genes candidatos implicados en la diplosporia, la partenogénesis y el desarrollo autónomo del endospermo. Realicé análisis bioinformáticos de datos de genotipado y secuenciación de alto rendimiento, incluyendo estudios de estructura y asociación de poblaciones, para descubrir regiones genómicas relacionadas con la apomixis. Se empleó citometría de flujo para evaluar los niveles de ploidía y el tamaño del genoma, lo que respaldó la clasificación fenotípica de las accesiones. Además, integré datos de metilación del ADN y secuenciación del ARN para detectar diferencias epigenéticas entre individuos apomícticos y sexuales. Estos enfoques integradores mejoraron nuestra comprensión de la base molecular de la apomixis, en consonancia con el objetivo del proyecto de aprovechar este rasgo para mejorar los cultivos.
Imagen: Viviana Echenique (izquierda) & Alessandro Mariani (derecha) en CERZOS.

Giacomo Bongiorno, PhD student
Universidad de Perugia, Italia
CERZOS (Bahia Blanca, Argentina), del 20 de septiembre 2024 al 21 de noviembre 2024
Durante esta comisión de servicio, realicé un análisis comparativo de las variaciones de presencia y ausencia (PAV) en Eragrostis curvula, que resultó en la identificación de 503 genes candidatos para la apomixis. Un subconjunto de ellos se validó mediante RT-PCR. También realicé llamadas de variantes SNPs e InDels, incorporando nuevos genotipos para investigar más a fondo la variación genómica, los modos reproductivos y los niveles de ploidía. El árbol filogenético construido utilizando estos variantes clasificó los genotipos en dos ramas principales, distinguiendo los genotipos altamente poliploides de los diploides y tetraploides. Este análisis proporcionó una visión más profunda de las relaciones genéticas entre genotipos de diferentes niveles de ploidía, arrojando luz sobre nuevos escenarios relacionados con la evolución del genoma en E. curvula.
Imagen: Giacomo Bongiorno (izquierda) y José Carballo (dereche)

Ing. Caroline Michaud
IRD, Montpellier, Francia
IICAR, Rosario (Argentina), del 12 de noviembre al 13 de diciembre de 2024
Durante esta comisión de servicio, contribuí al trabajo de Luciana Delgado y Daphné Autran sobre Paspalum rufum, explorando los vínculos entre la arquitectura del primordio del óvulo y la plasticidad del destino celular que conduce a la aposporía. Para determinar los patrones de proliferación celular durante el desarrollo del óvulo en Paspalum rufum sexual y apomíctico en estadios pre-meióticos, realizamos experimentos de marcaje con EdU para marcar células en fase S del ciclo celular utilizando el kit Click-iT™ EdU. Los ovarios fueron disecados en cuatro estadios tempranos, procesados para la incorporación y detección de EdU, luego se tomaron imágenes utilizando un microscopio confocal ZEISS 880 en el Instituto de Biología Molecular y Celular (IBR-CONICET) de Rosario. Estas imágenes serán segmentadas con MorphographX (MGX) para cuantificar las tasas de proliferación diferenciales entre genotipos. También he colaborado en un proyecto de Olivier Leblanc y Lorena Siena, construyendo plásmidos para el análisis funcional del gen TGS1 en Arabidopsis thaliana, con finalización prevista en Montpellier en 2025.
Imagen. Izquierda arriba : mi credencial de ¡docente de la UNR! ; Izquierda abajo : el microscopio confocal del IBR; Derecho : un diagrama de los estadios disecados de Paspalum rufum.

Dr. Olivier Leblanc
IRD, Montpellier, Francia
IICAR, Rosario (Argentina), del 12 de noviembre al 13 de diciembre de 2024
Imagen: Recorriendo la pampa desde Rosario hasta Bahía Blanca
Dr. Tamunonengiye-ofori Lawson
Cambridge, Reino Unido, NIAB
CERZOS (Bahia Blanca, Argentina), del 31 de octubre al 15 de diciembre de 2024

Dra. Daphné Autran
IRD, Montpellier, Francia
IICAR, Rosario (Argentina), del 28 de octubre al 31 de diciembre de 2024
Durante esta comisión de servicio, en el marco del WP6 del proyecto MAD, Daphné Autran (IRD) colaboró con Luciana Delgado (IICAR-CONICET), con la ayuda de Caroline Michaud (IRD), para descifrar la morfogénesis del óvulo en la especie apospórica Paspalum rufum. Para determinar cuáles son los eventos celulares más tempranos asociados con un programa de desarrollo apospórico facultativo, aprovechamos el período de floración de la población única de plantas contrastantes de P. rufum disponible en el IICAR, Zavalla, Rosario, Argentina. Aquí, se abordó específicamente el proceso de proliferación celular, monitoreando el número y localización de células en división, utilizando óvulos vivos, recolectados en diferentes estadios de desarrollo. Después de la fijación, limpieza y tinción, las muestras fueron visualizadas utilizando microscopía de fluorescencia confocal (disponible en el IBR, Instituto de Biología de Rosario). A continuación, se analizará el conjunto de datos de imágenes 3D generado, para completar la información recopilada del atlas de crecimiento celular 3D de óvulos de P. rufum producido en adscripciones anteriores.
Imagen: Luciana Delgado (izquierda) & Luciana Delgado y Daphné Autran (columna derecha)

Dra. Silvina Pessino
IICAR, Rosario, Argentina
Universidad de Milano, del 2 de octubre al 27 de noviembre de 2024
La Dra. Silvina Pessino (IICAR-CONICET, Argentina) fue destacada a la UMIL, Italia, del 2 de octubre al 27 de noviembre de 2024, para investigar el papel de varios FACTORES RESPONSABLES A LA AUXINA (ARFs) y sus respectivos controladores miRNA en la especificación de la línea germinal femenina en Arabidopsis thaliana, en colaboración con la Dra. Lucia Colombo y la Dra. Marta Mendes. En particular, durante esta comisión de servicio, participó en los pasos finales de publicación del artículo Pessino SC et al. (2024) AUXIN RESPONSIVE FACTOR 10 insensitive to miR160 regulation induces apospory-like phenotypes in Arabidopsis. iScience; preparó varios entregables del proyecto MAD y analizó datos McSeED y RNA-seq que apuntan al control de la expresión de ARF mediado por el sistema RdDM.
Imagen: Marta Mendes (izquierda) & Silvina Pessino (derecha)

Dr. Juan Pablo A. Ortiz
IICAR, Rosario, Argentina
Universidad de Padova, del 1ro de octubre 2024 al 1ro de noviembre 2024
El Dr. Juan Pablo A. Ortiz realizará una nueva comisión de servicio para el análisis de secuencias relacionadas con la apomixis, con especial atención en el descubrimiento de genes asociados a la partenogénesis. El trabajo incluirá el análisis de las bases de datos transcriptómicos de genotipos sexuales y apomícticos para identificar genes relacionados con cascadas hormonales, procesamiento de ARN y señalización celular que puedan estar implicados en la partenogénesis. El análisis también incluirá los recursos genómicos de locus controlador de la apomixis ya disponibles. Este trabajo se llevará a cabo en colaboración con el Dr. Giovanni Gabelli del grupo DAFNAE. Los resultados obtenidos contribuirán al WP3 y WP4 y pueden aportar nuevos candidatos de la inducción de la partenogénesis en especies apomícticas naturales.
Foto: Tomando maté… de izquierda a derecha: Juan Pablo Ortiz, Lorena Siena & Samela Draga

Dra. Lorena Siena
Rosario, Argentina, IICAR
IBBR (Italia), del 30 de agosto 2024 al 29 de septiembre 2024
Durante su vista al laboratorio del Dr. Fulvio Pupilli, Lorena trabajará en experimentos de secuenciación de plantas tetraploides sexuales y apomícticas de Paspalum simplex con el fin de mejorar los ensamblajes de genomas tetraploides disponibles. La información producida contribuirá al WP3 y se utilizará para estudiar la estructura y evolución del genoma, así como, para descifrar el contenido génico del locus de control de la apomixis. Este trabajo se llevará a cabo en colaboración con el Dr. Andrea Rubini y el Lic.Davide Perrone del IBBR y el Dr. Juan Pablo A Ortiz del IICAR. Además, Lorena trabajará en la puesta a punto de ensayos de hibridación in situ para analizar el patrón de expresión del gen candidato ARF5 de P. simplex en ovarios apomícticos y sexuales. Participará en el diseño y marcado de la sonda, y en el montaje en parafina de los ovarios. El seccionamiento y la hibridación se llevarán a cabo en el IICAR, CONICET-UNR. Estos experimentos se realizarán en colaboración con la Dra Eugenia Cáceres del IBBR.
Imagen: Lorena Siena (derecha) & Davide Perrone (izquierda)

Dr. Juan Pablo A. Ortiz
Rosario, Argentina, IICAR
IBBR (Italia), del 30 de agosto 2024 al 29 de septiembre 2024
El objetivo de esta comisión de servicio es profundizar la caracterización de los genomas sexual y apomíctico de Paspalum simplex. El plan de trabajo incluye la extracción de ADN, la preparación de bibliotecas y los experimentos de secuenciación de genotipos tetraploides utilizando la tecnología ONT. Estas actividades contribuirán al WP3 cuyo objetivo es analizar la estructura y evolución de los genomas apomícticos y dilucidar el contenido génico y la función del locus de la apomixis. Los otros miembros del equipo que participarán en este trabajo son la Dra. Lorena Siena (IICAR), el Dr. Andrea Rubini (IBBR) y el BSc Davide Perrone (IBBR). Como parte de la visita, Juan Pablo también trabajará en la construcción de un vector plasmídico de ARNi diseñado para regular a la baja el gen ORC3, uno de los candidatos identificados por el grupo del Dr. Pupilli en el IBBR como el principal regulador del desarrollo del endosperma en semillas apomícticas. La construcción del ARNi se utilizará para generar plantas transgénicas de maíz, como parte de una estrategia para validar su efecto en la formación de semillas con exceso de genoma materno en plantas sexuales. Esta parte del trabajo se realizará en colaboración con la Dra. Francesca De Marchis y el Dr. Michelle Bellucci, del IBBR.
Imagen: Juan Pablo A. Ortiz (izquierda) & Fulvio Pupilli (derecha)

Ing. Lopez Villegas
CERZOS, Bahia Blanca, Argentina
IRD (Francia) del 20 de junio 2024 al 26 de agosto 2024
El Ing. David López Villegas (Bahía Blanca, Argentina) realizó una estancia en el IRD de Montpellier, Francia, del 24 de junio al 23 de agosto de 2024. Durante su estancia, adquirió las competencias y los conocimientos necesarios para llevar a cabo la valorización tecnológica en colaboración con la Oficina de Transferencia Tecnológica del IRD, centrándose específicamente en la apomixis y sus posibles aplicaciones en la agricultura. Realizó una revisión sistemática de las patentes relacionadas con la apomixis y las tecnologías asociadas, lo que le permitió obtener información valiosa sobre las tendencias tecnológicas actuales, las estrategias de propiedad intelectual y las principales instituciones y empresas activas en este campo.
Foto: David Lopez, IRD, Montpellier, Francia

Dr. Andrés Bellido
CERZOS, Bahia Blanca, Argentina
IRD (Francia) del 31 de mayo 2024 al 29 de junio 2024
Se generaron plantas transgénicas de Arabidopsis thaliana expresando genes relacionados con la apomixis y identificados en Eragrostis curvula. Estas plantas muestran varios fenotipos relacionados con la apomixis durante la especificación de la CMM y el desarrollo del saco embrionario, así como signos de pérdida de metilación del ADN. El objetivo de este trabajo es caracterizar las marcas histónicas más relevantes durante el desarrollo del óvulo en estas líneas transgénicas de Arabidopsis mediante técnicas de inmunomarcaje y microscopía confocal.

Dra. Viviana Echenique
CERZOS, Bahia Blanca, Argentina
Universidad de Perugia (Italia) del 30 de mayo 2024 al 2 de agosto 2024
El objetivo de esta comisión de servicio en UNIPG es continuar con el grupo del Dr. Albertini el trabajo para identificar la región genómica implicada en la apomixis en Eragrostis curvula y caracterizar su contenido genico. Para ello, analizaremos los datos obtenidos previamente del pangenoma de E. curvula, que consiste en la secuenciación Illumina de10 genotipos con genómicamente similares pero con diferentes modos reproductivos (apomíctico y sexual) y niveles de ploidía (2x, 4x, 6x, 7x y 8x). Los datos se utilizarán para estudiar las regiones genómicas implicadas en la apomixis y su sintenia con las homólogas sexuales mediante un enfoque cartográfico para obtener el primer ensamblaje del pangenoma de E. curvula.

Dra. Maria Cielo Pasten
CERZOS, Bahia Blanca, Argentina
Universidad de Milano (Italia) del 30 de mayo 2024 al 2 de agosto 2024
El objetivo de esta comisión de servicio era iniciar la caracterización de líneas mutantes SALK en Arabidopsis thaliana para los genes CHC2 (Clathrin heavy chain 2) y NFD3 (Nuclear fusion defective 3), ambos identificados previamente como expresados diferencialmente en genotipos apomícticos y sexuales de Eragrostis curvula. Se llevó a cabo el genotipado con cebadores específicos y actualmente se están estudiando varias de las líneas heterocigóticas identificadas. También hemos obtenido plantas mutadas mediante CRISPR-Cas9 para el gen SPL7 (Squamosa promoter binding like 7). Actualmente se están caracterizando. Además, se llevó a cabo una hibridación in situ (ISH) para determinar el perfil de expresión del gen SPL7 en los tejidos reproductivos de individuos diploides y tetraploides de A. thaliana (ecotipo Col0).

Domenico Loperfido, MSc
Universidad de Milano, Italia
CERZOS (Bahia Blanca, Argentina) del 11 de marzo 2024 al 8 de junio 2024
Durante los tres meses (del 11 de mayo al 8 de junio de 2024) que pasó en CERZOS, Domenico Loperfido trabajó en el laboratorio del Prof. Echenique bajo la supervisión de José Carballo y Cristian Gallo del CERZOS y del Prof. Ezquer de la Universidad de Milán. El objetivo de la comisión de servicio era investigar genes candidatos implicados en el crecimiento y desarrollo de semillas mediante estudios de asociación encaminados a entender el papel que estos genes pueden tener en la herencia de procesos apomícticos. Se utilizaron genomas de diferentes accesiones de Arabidopsis thaliana ; utilizando herramientas bioinformáticas, por ejemplo bcftools, vcftools, R, GAPIT, y otras, se identificaron SNPs significativos en genes fenotípicamente asociados con el crecimiento y el desarrollo de los componentes de la semilla (embrión, endospermo y tegumentos) que controlan el vigor híbrido en plantas sexuales.

Dr. Diego Zappacosta
CERZOS, Bahia Blanca, Argentina
Universidad de Milano (Italia) del 5 de marzo 2024 al 5 de mayo 2024
El objetivo principal de esta comisión de servico es continuar con la caracterización de una población segregante para la apomixis en Eragrostis curvula con el fin de generar un mapa de ligamiento y seleccionar individuos con marcadores moleculares similares pero diferentes modo reproductivo (apomíctica y sexual) para futuros análisis transcriptómicos. Durante estos dos meses, en colaboración con el grupo UNIPG, se plantea continuar el análisis de los datos fenotípicos y genoticos de una población de mapeo de E. curvula para genenerar un nuevo mapa consenso e identificar marcadores moleculares relacionados con la apomeiosis y, en última instancia, descubrir genes que podrían desempeñar un papel en el control de la reproducción apomíctica. Además, también contribuiré a la elaboración de un calendario destinado a asociar parámetros morfológicos florales con las fases de desarrollo de la megasporogénesis y la megagametogénesis para diferentes genotipos apomícticos y sexuales en E. curvula (Tarea 3.3).
Lucia Giordano, doctorante
Universidad de Perugia, Italia
Universidad de Adelaide (Australia), del 18 de enero 2024 al 2 de junio 2024
2023
Samela Draga, estudiante de doctorado
Universidad de Padova, Italia
IICAR (Rosario, Argentina), del 3 de noviembre 2023 al 19 de enero 2024
El objetivo general de la visita de Samela Draga al laboratorio del Prof. Pessino del 3 de noviembre de 2023 al 18 de enero de 2024 es continuar el proyecto iniciado en marzo-julio de 2023 en el IICAR-CONICET-UNR, cuyo objetivo es comprender el papel del factor de splicing BUD13 (un interactor de TGS1) en plantas apomícticas y sexuales de Paspalum. BUD13 se expresa de forma diferencial en las flores de plantas sexuales y apomícticas en al menos dos especies apospóricas, Paspalum notatum e Hypericum perforatum. Curiosamente, los mutantes de bud13 en Arabidopsis están asociados a defectos en el desarrollo embrionario temprano. Durante su visita, Samela producirá una sonda BUD13 marcada con digoxigenina, preparará secciones de ovarios embebidos en bloques de parafina y realizará análisis ISH. Las observaciones permitirán determinar los perfiles de expresión espacial de BUD13 en los órganos reproductores de los genotipos sexual y apomíctico de Paspalum notatum.
Dr. Giovanni Gabelli
Universidad de Padova, Italia
IICAR (Rosario, Argentina), del 15 de noviembre 2023 al 15 de diciembre 2023

Dr. Fulvio Pupilli
IBBR, Perugia, Italia
IICAR (Rosario, Argentina), del 8 de noviembre 2023 al 9 de dicembre 2023
Durante su primera comisión de servicio en Rosario, del 9 de noviembre al 14 de diciembre de 2023, Fulvio Pupilli discutió con Juan Pablo Ortiz, su equipo y Olivier Leblanc los recientes avances en secuenciación Nanopore y el ensamblaje de genomas apomícticos y sexuales en Paspalum simplex. Además, se discutieron con JP Ortiz los progresos realizados en la caracterización del gen PsARF5, que se expresa de forma diferencial entre los tejidos nucelares disecados por LCM de genotipos apomícticos y sexuales, así como las posibles interacciones con PnIAA30, un gen que actualmente se está caracterizando funcionalmente en el IICAR. Para la próxima comisión de servicio se ha programado una serie de experimentos de hibridación in situ para PsARF5. Por último, ha preparado una presentación sobre el papel del gen PsORC3 en el desarrollo del albumen con exceso genómico materno en P. simplex y un homenaje al profesor Camilo Luis Quarin para el IV congreso Apomixis, que se celebrará del 3 al 7 de diciembre de 2023 en Rosario, Argentina.

Giacomo Bongiorno, estudiante de doctorado
Universidad de Perugia, Italia
CERZOS (Bahia Blanca, Argentina), del 18 de mayo 2023 al 9 de dicembre 2023
Durante esta comisión de servicio en CERZOS, me centré principalmente en la gramínea diplospórica Eragrostis curvula con el objetivo de construir un pangenoma completo. El uso del cultivar Victoria como genoma de referencia proporcionó una base sólida para estudiar la variación genómica, las relaciones filogenéticas y el impacto de la ploidía en la evolución del genoma y los patrones de reproducción. Este estudio pangenómico supone una contribución significativa a nuestra comprensión de la diversidad genómica de E. curvula, como recurso de interés para investigaciones específicas sobre los mecanismos moleculares que rigen sus modos reproductivos y sus relaciones con la ploidía.
Foto (de izquierda a derecha) : Juan Pablo Selva, Diego Zappacosta, Juan Manuel Rodrigo, Nengi Lawson, Viviana Echenique, Alejandra Diaz & Giacomo Bongiorno

Dr. Daphné Autran
IRD, Montpellier, France
IICAR (Rosario, Argentina), del 22 de octubre 2023 al 17 de diciembre 2023
Durante esta comisión de servicio, seguimos trabajando en Paspalum rufum con Luciana Delgado. Para comprender las interrelaciones entre la arquitectura del primordio del óvulo y la plasticidad del destino celular que conduce a la aposporía, estamos realizando un análisis morfométrico celular en 3D de genotipos de P. rufum que muestran una expresividad diferencial de la aposporía. El primer paso es construir un atlas 3D de referencia del genotipo sexual. Tras el trabajo de segmentación, etiquetado y exportación de datos realizado en las sesiones anteriores mediante el software MorphographX (MGX), nos dispusimos a realizar un análisis cuantitativo en un grupo piloto de 11 óvulos en estadios sucesivos que cubrían la ventana de desarrollo de interés. Además de la cuantificación del número y tamaño de las células, se seleccionaron diferentes descriptores de forma, tanto a nivel celular como de órgano, y se calcularon a partir de las características extraídas por MGX. Dado que los datos piloto ya abarcan miles de células para analizar, se escribieron scripts personalizados en R para gestionar la compilación de datos, la visualización y el análisis estadístico. Además, se diseccionaron, fijaron y limpiaron óvulos de P.rufum en torno a la fase de meiosis, para ampliar la ventana de desarrollo cubierta y maximizar la probabilidad de detección de crecimiento celular diferencial entre genotipos sexuales y apospóricos.
Paspalum rufum, Luciana Delgado & Daphné Autran

Dr. Olivier Leblanc
IRD, Montpellier, France
IICAR (Rosario, Argentina), del 15 de octubre 2023 al 13 de diciembre 2023
Esta segunda visita al IICAR forma parte de una comisión de servicio dedicada a la genómica de la apomixis (WP3) y a la caracterización funcional de los genes candidatos de Paspalum para la apomixis utilizando mutantes de Arabidopsis (WP4). En cuanto a los enfoques genómicos, el primer objetivo es finalizar la redacción de un primer artículo que informara del ensamblaje y la anotación detallada de un genoma diploide de referencia de P. notatum. Además, centraremos nuestros análisis en la comprensión de las variaciones entre los genomas de las plantas diploides sexuales y las plantas tetraploides apomícticas utilizando enfoques de genómica comparativa y k-mers. En cuanto a la caracterización funcional de los mutantes de Arabidopsis, se analizarán los efectos de las mutaciones de tgs1 en el corte y empalme del ARN mediante análisis transcripcionales comparativos y herramientas citológicas (reporteros fluorescentes en desarollo). También estoy organizando el segundo taller ONT MAD sobre secuenciación y bioinformática (Rosario, 20-25 de noviembre) y miembro del comite organizador de la conferencia Apomixis2023 (Rosario, 3-7 de diciembre).
Imagen: visitando a viejos conocidos en Cayasta (Santa Fe, Argentina), cuna de la familia Paspalum. de izquierda a derecha: O Leblanc, JPA Ortiz, & L Siena

Davide Perrone
Universidad de Milán, Italia
IICAR (Rosario, Argentina), del 12 de septiembre 2023 al 13 de diciembre 2023
Davide Perrone está visitando el IICAR-CONICET para continuar los experimentos iniciados durante una comisión de servicio anterior, relativos a la transformación de plantas apomícticas y sexuales de Paspalum notatum con reporteros fluorescentes de auxina y citoquinina. El objetivo de estos experimentos es comparar la distribución de hormonas en los óvulos de plantas sexuales y apomícticas. Además, Davide iniciará experimentos de hibridación in situ destinados a comparar la expresión del gen AUXIN RESPONSE FACTOR 10 (ARF10) en los óvulos de los biotipos de biotipos sexuales y apomícticos de Paspalum notatum. Por último, en colaboración con científicos del IICAR, llevará a cabo análisis miR-seq utilizando datos generados previamente en Milán.
Foto (de izquierda a derecha): Augustus Villalba (IBONE), Silvina Pessino (IICAR), Maricel Podio (IICAR) & Davide Perrone (Univ. Milán)
Dr. Cristian Gallo
Bahia Blanca, Argentina, CERZOS
Universidad de Perugia (Italia), del 1 de septiembre 2023 al 30 de octubre 2023

Dra. Francesca De Marchis
Perugia, Italia, CNR-IBBR
IICAR (Rosario, Argentina), del 8 de agosto 2023 al 12 de septiembre 2023
Francesca De Marchis es investigadora del Consejo Nacional de Investigación, CNR- IBBR – Perugia. Durante su visita, trabajará con el Dr. Juan Pablo A. Ortiz del IICAR – CONICET (Rosario, Argentina) y su experiencia en biotecnología vegetal será aprovechada para diseñar un vector para la transformación de Paspalum notatum. Algunos genes candidatos para la partenogénesis, identificados por el grupo de investigación del IICAR, serán considerados para la transformación de plantas mediante tecnología biolística eligiendo la sobreexpresión o la represión.

Dra. Alejendra Díaz
Bahia Blanca, Argentina, CERZOS
NIAB (Reino Unido), del 1 de agosto 2023 al 2 de octubre 2023
Nuestro grupo de investigación CERZOS lleva varios años colaborando con el grupo del Dr. Caccamo en el NIAB para identificar y caracterizar una región genómica implicada en la apomixis en Eragrostis curvula (Schrad.) Nees, así como sus genes. Algunos de estos genes, previamente identificados por nuestro grupo de investigación como implicados en la apomixis y para los que no se dispone de anotación en otras especies, están siendo actualmente caracterizados funcionalmente. La expresión de estos candidatos bajo promotores constitutivos en la planta modelo, el arroz, es una de las vias utilizadas para caracterizarlos. El objetivo de esta comisión de servicio en el NIAB era avanzar en la caracterización de uno de nuestros genes candidatos, principalmente para determinar si su sobreexpresión induce defectos en el desarrollo del saco embrionario. Para ello, participé en el cribado de tejidos reproductivos de espiguillas de líneas de arroz transgénico independientes generadas en el NIAB y que sobreexpresaban el gen de interés (WP4). Utilizamos microscopía de contraste de interferencia diferencial (DIC) para observar tejidos reproductivos aclarados con una solución de salicilato de metilo, ya que esta técnica permite realizar observaciones a gran escala de la meiosis y el desarrollo del saco embrionario en ovulos intactos. La colaboración entre el NIAB y CERZOS nos permitirá caracterizar más rápidamente los genes candidatos a la apomixis identificados en Eragrostis.

Dra. Viviana Echenique
Bahia Blanca, Argentina, CERZOS
NIAB (Reino Unido), del 1 de agosto 2023 al 2 de octubre 2023
El objetivo de mi comisión de servicio en el NIAB era continuar con el trabajo iniciado con el grupo del Dr. Caccamo relacionado con la identificación y caracterización de una región genómica y de genes implicados en la apomixis. Aquí discutimos estrategias y diseñamos nuevos experimentos. El trabajo en curso consiste en la secuenciación y ensamblaje del genoma del cultivar apomíctico Don Walter (Eragrostis curvula). Este genoma fue secuenciado en el NIAB usando la plataforma Oxford nanopore con el protocolo de modelo de planta de super-alta precisión y uno de los estudiantes del CERZOS está trabajando aquí haciendo el ensamblaje. También estamos involucrados en un proyecto de transcriptómica utilizando datos de RNA-seq obtenidos de tejidos de pistilo de plantas apomícticas y sexuales en tres etapas de desarrollo diferentes. Estamos buscando genes expresados diferencialmente entre plantas con diferentes modos reproductivos y diferentes estados de desarrollo.
Foto: Viviana en The Eagle, Cambridge (Inglaterra)

Sol Vega, estudiante de doctorado
Rosario, Argentina, IICAR
IRD (Francia), del 20 de julio 2023 al 20 de septiembre 2023
En esta comisión de servicio María Sol visita el IRD para continuar con la caracterización funcional del gen IAA16 en Arabidopsis thaliana, cuyo ortólogo (IAA30) se ha asociado con la apomixis en Paspalum notatum. Las actividades se llevarán a cabo en el laboratorio del Dr. Olivier Leblanc e incluirán la selección de plantas dobles homocigóticas derivadas de cruzas entre la mutante iaa16 y; una línea portadora del reportero gametofítico pWOX2-CENH3-GFP; plantas tgs1 (relacionadas con alteraciones en el desarrollo gametofítico) y; plantas topless (implicadas en la respuesta a la auxina durante la embriogénesis). Los análisis fenotípicos del desarrollo reproductivo del material obtenido se llevarán a cabo mediante microscopía de fluorescencia o DIC durante la visita al IRD y posteriormente en el IICAR. Además, se iniciará el estudio de mutantes para otros genes del clado IAA16 (iaa14 y iaa17) así como para una nueva inserción del gen IAA16.

Dra. Lorena Siena
Rosario, Argentina, IICAR
IRD (Francia), del 22 de junio al 16 de septiembre 2023
En un nuevo secondment, Lorena Siena, del IICAR, está visitando el IRD por tres meses. El objetivo de la visita es avanzar en la caracterización funcional de los genes TGS1 y QGJ durante el desarrollo reproductivo de Arabidopsis thaliana. Las actividades se llevarán a cabo en colaboración con el grupo del IRD y, en primer lugar, incluyen el análisis del potencial de embriones partenogenéticos en líneas mutantes tgs1 mediante el uso de reporteros fluorescentes específicos de embrión transmitidos maternal y paternalmente. Además, se caracterizará ampliamente la identidad celular de las megasporas adicionales y funcionales observadas en los óvulos qgj, por ejemplo utilizando el reportero pWOX2-CENH3-GFP. Por otra parte, iniciaremos experimentos destinados a identificar posibles interacciones entre QGJ y TGS1 determinando el patrón de expresión de QGJ (pQGJ-GUS) en óvulos tgs1 y, recíprocamente, el de TGS1 (pTGS1:TGS1-GUS) en óvulos qgj.
Imagen: Aquaducto de Montpellier (@ Lorena Siena, IICAR)

Dr. Jose Carballo
Bahia Blanca, Argentina, CERZOS
NIAB (Reino Unido), del 14 de junio al 14 de septiembre 2023
El objetivo de mi comisión de servicio en el NIAB era continuar con la secuenciación y el ensamblaje del genoma apomíctico de Eragrostis curvula. El genoma del genotipo Don Walter fue secuenciado en el instituto NIAB utilizando la plataforma Oxford nanopore con el protocolo de modelo de planta de super-alta precisión. Estas lecturas se ensamblaron utilizando el software Flye y se andamillaron con la tecnología Omni-C para obtener un ensamblaje a escala cromosómica. Este genoma se ensambló y anotó obteniendo los subgenomas A y B del alotetraploide Don Walter. Además, durante este tiempo en Cambridge, se analizaron los datos de RNA-seq obtenidos de tejidos de pistilo de plantas apomícticas y sexuales en tres estados de desarrollo diferentes obteniendo genes diferencialmente expresados. Esta comisión de servicio continúa con los esfuerzos de colaboración entre el NIAB y CERZOS-CONICET para desvelar las bases moleculares de la apomixis a través de un enfoque bioinfirmatico.

Dr. Tamunonengiye-ofori Lawson
Cambridge, UK, NIAB
CERZOS (Bahia Blanca, Argentina), del 20 de mayo al 19 de julio 2023
El objetivo de esta estancia en el CERZOS era profundizar mi conocimiento, habilidades y experiencia práctica en el área de la hibridación in situ y la microscopía DIC en plantas.
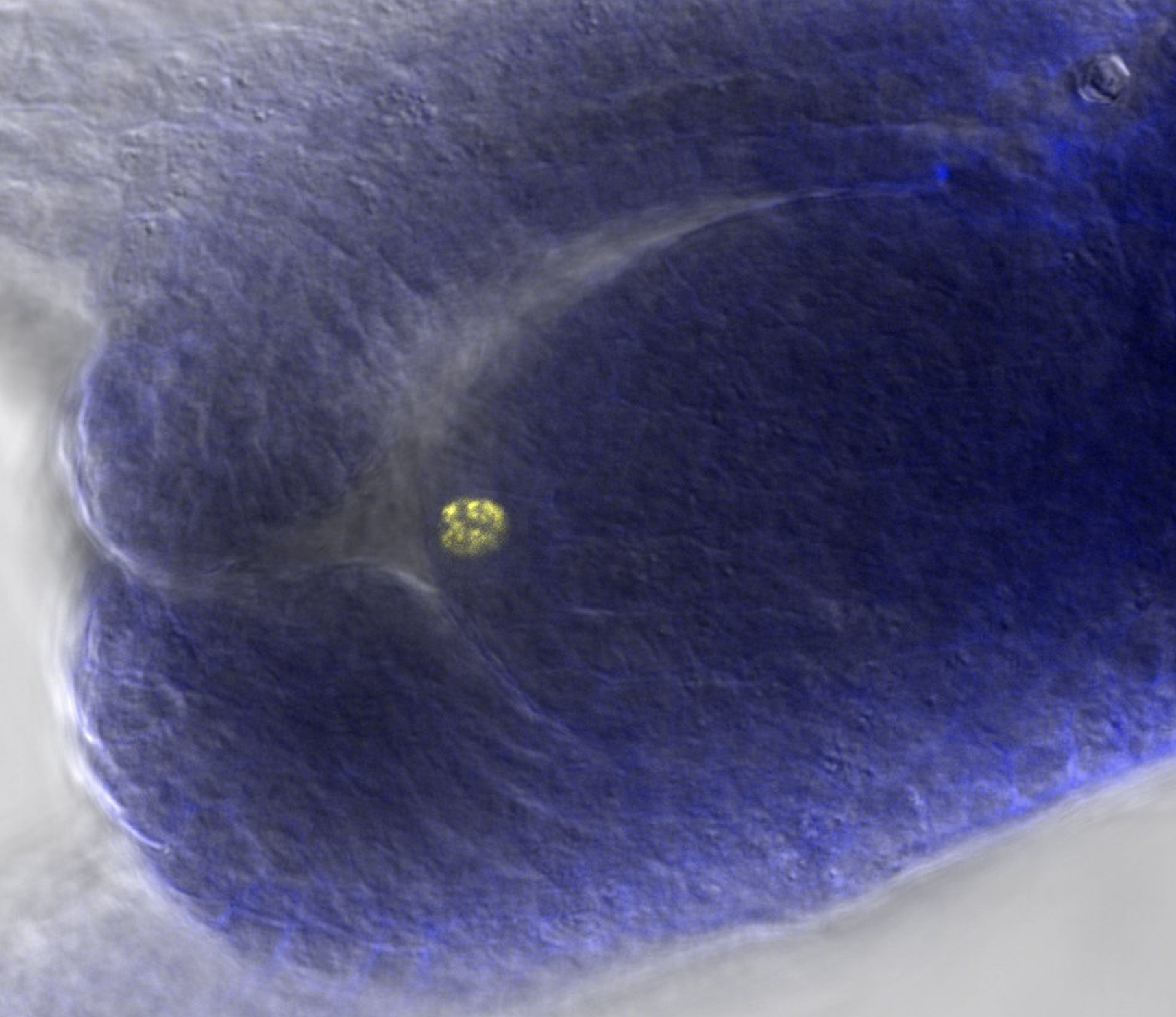
Dra. Luciana Delgado
Rosario, Argentina, IICAR
IRD (Francia), del 20 de mayo 2023 al 19 de agosto 2023
Esta comisión de servicio continua el trabajo iniciado en 2022 con Daphné Autran para producir un atlas 3D del desarrollo temprano del óvulo en Paspalum rufum. Realizaremos la segmentación y clasificación de células dentro de los óvulos utilizando imágenes de alta resolución obtenidas previamente y un procedimiento previamente establecido y parcialmente automatizado mediante un workflow utilizando el software MorphoGraphX. Además, probaremos diferentes protocolos para incorporar y detectar un análogo de la timidina (EdU) y cuantificar las divisiónes celulares durante el desarrollo de óvulos de diferentes gramíneas, incluyendo dos especies sexuales modelo (arroz y maíz) y plantas apomícticas de Paspalum. Estos protocolos no fueron aun optimizados para ovulo de gramíneas con disparidades morfológicas en flores y óvulos. La información cuantitativa así recogida del atlas 3D y de las divisiónes celulares en óvulos de plantas sexuales y apomícticas mejorarà nuestra comprensión de la transición entre sexualidad y apomixis.
Imagen : Célula interfásica en un óvulo joven de Paspalum rufum con señal fluorescente del EdU incorprado al ADN.

Samela Draga, estudiante de doctorado
Universidad de Padova, Italia
IICAR (Rosario, Argentina), del 16 de mayo 2023 al 8 de agosto 2023
El objetivo general del trabajo de Samela en IICAR es contribuir a la elucidación del papel de la proteína de splicing BUD13 en la transición sexualidad-apomixis. En particular, caracterizará BUD13 en las bases de datos transcriptómicas y genómicas de Paspalum notatum y generará sondas para su detección mediante hibridación in situ de ARN. Además, clasificará los óvulos de plantas sexuales y apomícticas de Paspalum notatum según las etapas de desarrollo mediante citoembriología DIC, los deshidratará en una serie de etanol/xilol y los incluirá en parafina. En una segunda visita (octubre-diciembre), preparará secciones de óvulos para hibridarlos con sondas de ARN sentido y antisentido marcadas con digoxigenina y detectará la señal DIG mediante reacción colorimétrica (anticuerpo DIG conjugado con fosfasa alcalina, sustratos NBT/BCIP). Esperamos obtener información sobre la expresión espacial de estos genes candidatos en genotipos apomícticos y sexuales.
Samela Draga (derecha) & Silvina Pessino (izquierda)

Nicola Babolin, estudiante de doctorado
Universidad de Milan, Italia
Universidad de Adelaide (Asutralia), del 10 de avril 2023 al 13 de octubre 2023
El objetivo de esta comisión de servicio en la Universidad de Adelaida (Australia) en el laboratorio A/Prof. Matthew Tucker es de caracterizar mejor la red molecular entre los dos genes MADS-box STK y ABS durante el proceso de fertilización y el desarrollo de semillas en Arabidopsis thaliana.
Al mismo tiempo participaré en los proyectos dirigidos por el prof. Tucker en relación con el óvulo y el desarrollo de semillas en cebada, que están muy interconectados con mi proyecto sobre Arabidopsis.
de izquierda a derecha : Nicola Babolin, Matthew Tucker, Rosanna Petrella

Dra. Rosanna Petrella
Universidad de Milan, Italia
Universidad de Adelaide (Asutralia), del 10 de avril 2023 al 13 de octubre 2023
Durante mi comisión de servicio en la Universidad de Adelaida (laboratorio de Matthew Tucker) mi trabajo se centrará en desentrañar los mecanismos moleculares que determinan una correcta progresión de la línea germinal femenina en Arabidopsis thaliana. En particular, intentaré caracterizar los principales actores implicados en la degeneración de las esporas y en la selección y diferenciación funcional de las megasporas al final de la megasporogénesis en el óvulo. Determinaré la contribución de la interacción fitohormonal intentando rescatar mutantes defectuosos en estos procesos clave utilizando regiones reguladoras específicas activas en diferentes dominios del óvulo para dirigir específicamente la expresión de genes candidatos.
de izquierda a derecha : Nicola Babolin, Skippy el canguro, Rosanna Petrella

Mattia Spano, estudiante de doctorado
Universidad de Milan, Italia
LANGEBIO (Mexico), del 1ro de abril 2023 al 30 de julio 2023
El objetivo de esta comisión de servicio es investigar el mecanismo molecular que determina la línea germinal femenina. En particular, investigaré las contribuciones de las fitohormonas, auxina y citoquininas, en la diferenciación de la MMC. Las especies analizadas son Arabidopsis thaliana y Oryza sativa var. Kitaake. En este marco, realizaré ensayos de inmunolocalización en diferentes fondos para localizar las fitohormonas durante el desarrollo del óvulo utilizando mutantes spl/nzz y de tipo salvaje para Arabidopsis y de tipo salvaje para Oryza sativa.

Dr. Juan Pablo A. Ortiz
Rosario, Argentina, IICAR
IRD (Francia), del 7 de marzo 2023 al 3 de abril 2023
El pasado 7 de marzo de 2023 se inició una nueva estancia del Dr. Juan Pablo A. Ortiz del IICAR en el IRD, en el marco de una comisión de servicio del MAD para caracterizar los genomas apomícticos (WP3). El plan de trabajo incluye la extracción de ADN de varias muestras de Paspalum spp para su secuenciación con Illumina y el análisis estructural del genoma. La visita también está dedicada a la preparación de un nuevo manuscrito que describe la secuenciación, el ensamblaje con resolución a nivel cromosómico y la anotación del genoma diploide de Paspalum notatum. Este resultado, fruto de un esfuerzo de colaboración entre diferentes socios argentinos, italianos y franceses del proyecto MAD, es un paso clave para el progreso del WP3 ya que servirá de referencia para caracterizar los genomas tetraploides y determinar la estructura y evolución de la región implicada en la apomixis. Por último, durante esta visita se discutirá y decidirá el programa del taller internacional sobre secuenciación por ONT y análisis bioinformático organizado por el proyecto MAD en Rosario, Argentina, para el próximo mes de noviembre.
Invernaderos del IRD. Juan Pablo A Ortiz (derecha) & Olivier Leblanc (izquierda)

Dra. Alejandra Díaz
Bahia Blanca, Argentina, CERZOS
Universidad de Milan (Italia), del 21 de enero 2023 al 23 de marzo 2023
El objetivo de esta comisión de servicio fue analizar la expresión tejido-específica de dos genes candidatos implicados en el modo reproductivo apomíctico en la gramínea Eragrostis curvula (WP4). Para ello, utilizamos la técnica de hibridación in situ de ARN (RNA-ISH) para determinar los patrones de expresión de estos dos genes en células de órganos reproductores de E. curvula, tanto en el genotipo apomíctico completo como en el sexual. Además, también caracterizamos un gen que demuestra una expresión diferencial entre plantas sexuales y apomícticas de E. curvula y que también se expresa en la planta modelo A. thaliana. Para este último objetivo, también utilizamos ISH para comprobar la abundancia de ARN y la localización de este gen ortólogo en los tejidos reproductivos de E. curvula (genotipos apomíctico y sexual) y A. thaliana (genotipos diploide y tetraploide).
de izquierda a derecha: Marta Mendes, Lucia Colombo, Alejandra Díaz y Cielo Pasten
2022
Gregorio Orozco, estudiante de doctorado
Universidad de Milan, Italia
LANGEBIO (Mexico), del 7 de diciembre 2022 al 3 de febrero 2023
Dra. Lucia Colombo
Universidad de Milan, Italia
IICAR (Rosario, Argentina), del 14 de noviembre 2022 al 27 de noviembre 2022
Jimena Gallardo
Bahia Blanca, Argentina, CERZOS
University of Perugia (Italia), del 2 de noviembre 2022 al 2 de diciembre 2022

Dr. Olivier Leblanc
Montpellier, France, IRD
IICAR (Rosario, Argentina), del 17 de octubre 2022 al 16 de diciembre 2022
Esta comisión de servicio tiene como objetivo (1) la caracterización de la región cromosómica asociada a la apomixis en forrajeras tropicales. Dado que ya hemos completado el ensamblaje y la anotación del genoma de un individuo diploide sexual de Paspalum notatum, se explorarán diferentes estrategias para revelar las variaciones estructurales que pueden explicar al desarrollo apomíctico en las especies del género. Para eso, hemos producido > 100 Gb de lecturas largas (tecnología Nanopore) de varias plantas apomícticas que se utilizarán para intentar ensamblar de novo estos genomas y realizar análisis comparativos, y; (2) profundizar el análisis funcional de mutantes tgs1 de Arabidopsis y, en particular realizar un análisis transcripcional comparativo entre embriones tgs1 y WT de Arabidopsis, colectados en etapas tempranas del desarrollo. Estas actividades forman parte del WP3 y del WP4, respectivamente.
Imagen: clones metálicos en la pampa rosarina, Provincia de Santa Fe, Argentina (@ O Leblanc)

Dra. Maria Cielo Pasten
Bahia Blanca, Argentina, CERZOS
Universidad de Milan (Italia), del 30 de septiembre 2022 al 1ro de marzo 2023
El objetivo de esta comisión de servicio era analizar los genes que muestran una expresión diferencial entre óvulos de plantas sexuales y apomícticas de Eragrostis curvula y que también están presentes en la planta modelo Arabidopsis thaliana. Para ello, estudiamos el fenotipo de mutantes de A. thaliana disponibles públicamente para algunos de estos genes, pero también el de nuevas líneas mutantes transgénicas generadas mediante la estrategia CRISPR-Cas9. Además, se caracterizó el desarrollo de los óvulos en genotipos apomícticos y sexuales de E. curvula mediante microscopía confocal, con el fin de conocer mejor las estructuras y las diferencias entre los modos de reproducción sexual y asexual.
de izquierda a derecha: Marta Mendes, Lucia Colombo, Alejandra Díaz y Cielo Pasten

Pr. Pablo Roncallo
Bahia Blanca, Argentina, CERZOS
Universidad de Perugia (Italia), del 6 de septiembre 2022 al 8 de noviembre 2022
El objetivo de esta comisión de servicio es ampliar un mapa de ligamiento genético tetraploide de Eragrostis curvula utilizando marcadores DarT seq y genotipos F1 adicionales con el fin de cartografiar con mayor precisión la región implicada en la apomixis. La población F1 se obtuvo cruzando un genotipo sexual (OTA) y uno apomíctico (Don Walter) como padres materno y paterno, respectivamente. Este nuevo mapa de ligamiento facilitará la localización de los loci que controlan la apomixis (diplosporia) y el estudio de la evolución del genoma de esta gramínea (WP3). En colaboración con el grupo de la UNIPG, estoy analizando los marcadores moleculares (DarT seq) obtenidos en los nuevos individuos F1 y en otros previamente obtenidos en Bahía Blanca por nuestro grupo para construir un nuevo mapa de ligamiento integrado, así como dos mapas de ligamiento específicos de los padres. Para ello, es necesario el análisis de segregación y la detección de marcadores SNP polimórficos en uno o ambos progenitores para resolver estos tres mapas de ligamiento. Además, se explorará la reconstrucción del haplotipo utilizando esta población F1 y el perfil SNP de los padres.

Davide Perrone
Universidad de Milan, Italia
IICAR (Rosario, Argentina), del 2 de septiembre 2022 al 28 de noviembre 2022
Davide Perrone (Universidad de Milán, Italia) está visitando el laboratorio de la Prof. Silvina Pessino en el IICAR CONICET, Rosario, Argentina, del 3 de septiembre al 27 de noviembre de 2022. Su trabajo, realizado en colaboración con la Dra. Carolina Colono (IICAR-CONICET-UNR), tiene como objetivo utilizar la transformación biolística para producir líneas fluorescentes de Paspalum notatum DR5 (marcador de auxina) y TCS (marcador de citoquinina) con diferentes modos reproductivos (sexualidad y apomixis aposporosa). Estos materiales vegetales serán útiles para estudiar los patrones hormonales en diferentes etapas de desarrollo del ovulo en un modelo apomíctico bien caracterizado. Además, Davide está contribuyendo a estudiar el papel del FACTOR DE RESPUESTA A LA AUXINA 10 (ARF10) en el transporte de la auxina dentro del óvulo de Arabidopsis, realizando caracterizaciones citoembriológicas de cruzas arf10 x PIN1 y arf10 x DII-Venus obtenidos previamente en el IICAR-CONICET-UNR. Finalmente, está involucrado en el análisis de la conexión funcional entre el FACTOR DE RESPUESTA A LA AUXINA 10 (ARF10) y la TRIMETHYL GUANOSINE SYNTHASE (TGS1) (un controlador de la aposporía reportado) en Paspalum.
de izquierda a derecha: Carolina Colono, Silvina Pessino, Davide Perrone

Sol Vega, estudiante de doctorado
Rosario, Argentina, IICAR
IRD (Francia), del 15 de junio 2022 al 16 de agosto 2022
Maria Sol est doctorante à l’IICAR-CONICET, Argentine, et elle visite le laboratoire du Dr. Olivier Leblanc à l’IRD (Montpellier, France). L’objectif principal de son séjour est d’initier l’analyse fonctionnelle de mutants d’Arabidopsis défectueux pour le gène IAA16, dont l’orthologue chez Paspalum notatum (PsIAA30) présente une réduction importznte de son expression pendant le développement apomictique (Siena et al., Plants 2022, 11, 472). Les mutants iaa16 hétérozygotes et homozygotes seront analysés par des observations cytoembryologiques afin de rechercher d’éventuels phénotypes reproducteurs femelles rappelant l’apomixie aposporique. Ce travail contribuera à déterminer les effets de la déplétion de IAA16 sur le développement reproductif d’Arabidopsis et à planifier une caractérisation fonctionnelle plus poussée de ce gène ou d’autres gènes appartenant à la même voie. Pendant sa visite, Maria Sol travaillera en collaboration avec le Dr Olivier Leblanc et Caroline Michaud de l’IRD, ainsi qu’avec Lorena Siena, sa directrice de thèse à l’IICAR.
Lorena Sierna (izquierda) & Sol Vega (derecha)

Dr. Juan Pablo A. Ortiz
Rosario, Argentina, IICAR
IRD (Francia), del 7 de junio 2022 al 7 de julio 2022
Una nueva comisión de servicio del MAD dirigida a trabajar en la caracterización de genomas apomícticos (WP3) comenzó el 7 de junio de 2022. En esta ocasión, el Dr. Juan Pablo A. Ortiz del IICAR (CONICET-UNR, Argentina) está visitando el laboratorio del Dr. Olivier Leblanc en el IRD (Francia). El plan de trabajo incluye la anotación de un ensamblaje del genoma de un ecotipo diploide de bahía de Pensacola (Paspalum notatum, var. saurae Parodi), obtenido previamente en una colaboración entre laboratorios de Francia, Italia y Argentina. También pretendemos identificar y caracterizar secuencias relacionadas con el locus controlador de la apomixis (ACL) en una raza tetraploide. En particular, utilizaremos la tecnología ONT de «secuenciación adaptativa» que permite la secuenciación de regiones de interés. Por lo tanto, esperamos enriquecer nuestra secuenciación con lecturas específicas de la planta apomíctica tetraploide y luego ensamblar el ACL. Olivier Leblanc, Cédric Mariac y Julie Orjuela (Unidad de investigación del DIADE), así como Juan Manuel Vega (MAD adscrito al IICAR) contribuirán a este trabajo.
Juan Pablo Ortiz (izquierda) & Olivier Leblanc (derecha)

Dra. Lorena Siena
Rosario, Argentina, IICAR
IRD (Francia), del 6 de junio 2022 al 1ro de septiembre 2022
Durante esta comisión de servicio de 3 meses, Lorena Siena (IICAR, CONICET- Argentina) está visitando el IRD con el objetivo de avanzar en la caracterización funcional del gen TGS1 durante el desarrollo reproductivo de la planta modelo Arabidopsis thaliana. Las actividades se llevarán a cabo en colaboración con el Dr. Olivier Leblanc y su grupo del IRD, e incluyen: ensayos de complementación utilizando la construcción pTGS1:TGS1-GUS; determinación del origen de los sacos embrionarios extra observados en los mutantes tgs1 utilizando líneas reporteras portadoras de los genes pWOX2 y CENH3 que informan tanto de la identidad gametofítica femenina como del número cromosómico (origen reducido vs. no reducido) , y; análisis de los datos de RNAseq obtenidos de embriones de tipo salvaje y tgs1 en etapas tempranas del desarrollo embrionario. Este estudio pretende determinar las alteraciones en los patrones de expresión génica causadas por la depleción de TGS1; identificar sus potenciales genes diana, y; explorar si el splicing alternativo controla la embriogénesis temprana en las plantas.
Olivier Leblanc (izquierda) & Lorena Siena (derecha):

Dra. Luciana Delgado
Rosario, Argentina, IICAR
IRD (Francia), del 20 de mayo 2022 al 19 de agosto 2022
En esta comisión de servicio, la Dra. Luciana Delgado (IICAR) y la Dra. Daphné Autran trabajan juntas para estudiar la comunicación entre la división, la expansión y la diferenciación celular a lo largo del desarrollo reproductivo femenino en P. rufum, una especie apomíctica. El objetivo general es construir un atlas 3D del desarrollo temprano del óvulo. Durante la estancia estableceremos las condiciones óptimas para la clarificación de los óvulos de P. rufum y las técnicas de tinción de la pared celular para obtener imágenes 3D de alta resolución. Las imágenes se segmentarán con MorphoGraphX, se clasificarán por etapas de desarrollo y, finalmente, se realizará un análisis cuantitativo de descriptores celulares. Creemos que la comprensión de la coordinación entre los procesos de desarrollo reproductivo con el crecimiento y la división celular, en un contexto apomíctico, podría ser esclarecedora para entender la regulación de la apomixis.
Daphné Autran (izquierda) & Luciana Delgado (derecha)

Dr. Juan Pablo Selva
Bahia Blanca, Argentina, CERZOS
University of Milan (Italia), del 16 de mayo 2022 al 16 de julio 2022
El objetivo de esta comisión de servicio fue continuar con la caracterización funcional de un gen expresado diferencialmente en plantas sexuales y apomícticas de Eragrostis curvula utilizando mutantes de maíz portadores de una inserción Mutator en el gen ortólogo.
Durante estos dos meses, colaboré con el grupo de la profesora Gabriella Consonni para evaluar diferentes poblaciones F2 de maíz que se plantaron en un campo experimental. Las plantas fueron genotipadas utilizando cebadores específicos para la inserción del transposón localizada en el primer exón del gen ortólogo y cebadores específicos para el alelo de tipo salvaje. Las actividades también incluyeron la observación de pistilos clarificados bajo el microscopio en busca de anormalidades durante el desarrollo reproductivo femenino.

Dr. Juan Pablo A. Ortiz
Rosario, Argentina, IICAR
IBBR (Italia), del 5 de mayo 2022 al 6 de junio 2022
El 9 de mayo comienza una nueva comisión de servicio del MAD de un mes de duración en la que participan investigadores de Argentina e Italia. En efecto, el Dr. Juan Pablo A. Ortiz del IICAR, (CONICET-UNR) está visitando el laboratorio del Dr. Fulvio Pupilli en el Instituto de Biociencias y Biorecursos (IBBR-CNR) de Perugia. El plan de trabajo se centra en el análisis funcional de ORC3, un gen que se segrega completamente ligado a la apomixis, en la formación del endospermo de semillas apomícticas de Paspalum simplex. Además, también se pretende avanzar en el ensamblaje y la caracterización del genoma de P. simplex utilizando lecturas de secuencias largas generadas con la tecnología Oxford Nanopore. Los miembros del grupo del Dr. Pupilli que participan en este proyecto son: Dra. María Eugenia Cáceres, Dr. Andrea Rubini, Dra. Francesca De Marchis y Dr. Michele Bellucci. Al final de la comisión de servicio, se espera haber completado la redacción de un manuscrito sobre el análisis funcional de PsORC3 y producido un borrador del ensamblaje del genoma de P. simplex.
Fulvio Pupilli (izquierda) & Juan Pablo A. Ortiz (derecha)

Dra. Carolina Colono & Prof. Silvina Pessino
Rosario, Argentina, IICAR
University of Milan (Italia), del 29 de abril 2022 al 2de julio 2022
Del 29 de abril al 2 de julio de 2022 la Dra. Carolina Colono y la Prof. Silvina Pessino visitarán el laboratorio de la Prof. Lucia Colombo en la Universidad de Milán, para explorar el papel del control hormonal en la transición de la sexualidad a la apomixis utilizando la planta modelo Arabidopsis thaliana.
De izquierda a derecha: Carolina Colono, Silvina Pessino y Lucia Colombo

Juan Manuel Vega, estudiante de doctorado
Rosario, Argentina, IICAR
IRD (Francia), del 19 de abril 2022 al 18 de junio 2022
Esta comisión de servicio forma parte del esfuerzo emprendido por el proyecto MAD (ver objetivos del WP3) para determinar la naturaleza, la evolución y el papel funcional de la región de control de la aposporía (ACR) en Paspalum notatum. Aprovechando un genoma diploide de referencia y conjuntos de datos de lecturas largas disponibles para apomícticos tetraploides, primero utilizaré el método “adaptative sampling” que ofrece la plataforma ONT para enriquecer la secuenciación de lecturas largas de ACR y luego realizaré un ensamblaje de alta cobertura tanto para un apomíctico obligado como para un apomíctico facultativo. También se aplicarán estudios complementarios mediante el uso de métodos open source destinados a identificar y filtrar las variaciones estructurales. En conjunto, esto debería permitir estudios de genómica estructural y comparativa para determinar el contenido genico y la capacidad de codificación/regulación dentro del ACR y proporcionar pistas sobre su papel funcional en la aparición de la apomixis en Paspalum.

Dr. Diego Zappacosta
Bahia Blanca, Argentina, CERZOS
University of Perugia (Italia), del 1 de marzo 2022 al 7 de mayo 2022
El objetivo de esta comisión de servicio es continuar con el ensamblaje de los genomas tetraploides de Eragrostis curvula para determinar el locus que controla la apomixis y estudiar la evolución del genoma en esta gramínea (WP 3). Durante estos dos meses, analizaré, en colaboración con el grupo de la UNIPG, las relaciones genéticas entre el diploide sexual (Victoria), previamente secuenciado y publicado por las 2 instituciones, y el tetraploide apomíctico cv. Tanganyika. Primero ensamblaremos un pan-genoma y lo resolveremos en el genoma central y el genoma prescindible. Se ensamblarán las lecturas mapeadas en la referencia Victoria, mientras que las lecturas no mapeadas se ensamblarán de novo para analizar las variaciones de presencia/ausencia. A continuación, analizaremos los genes ortólogos para encontrar contracciones y expansiones de familias de genes y compararemos las regiones que controlan la apomixis en Eragrostis y Paspalum spp. Además, aplicaré el protocolo MCSeEd desarrollado por UNIPG para comparar las regiones metiladas diferencialmente entre los genotipos apomícticos y sexuales (Task 3.3).
