Personnel détaché
La période de détachement est la période passée par un membre du projet dans un institut hébergeant, aux fins du projet. La personne détachée est le membre hébergé.
2025
Dr. Fulvio Pupilli
IICAR, Rosario, Argentine
IICAR (Rosario, Argentine), du 11 septembre au 12 novembre 2025

Dr. Viviana Echenique
CERZOS, Bahia Blanca, Argentine
Université de Pérouse (Italie), du 1er octobre au 30 octobre 2025
Photo: Viviana Echenique à l’UNIPG

Juan Manuel Vega, doctorant
IBBR, Pérouse, Italie
IBBR (Pérouse, Italie), du 11 septembre au 12 novembre 2025
Juan Manuel Vega, du groupe DREP de l’IICAR-CONICET (Rosario, Argentine), effectuera un détachement à l’IBBR-CNR (Pérouse, Italie) au sein du groupe du Dr Fulvio Pupilli du 11 septembre au 12 novembre 2025. Au cours de ce séjour, il travaillera à l’amélioration d’un assemblage préliminaire du génome de Paspalum simplex en réalisant un échafaudage basé sur le génome de référence de P. notatum. Il contribuera également à l’annotation des génomes sexuels et apomictiques de P. simplex et à l’identification et la caractérisation du locus de contrôle de l’apomixie (ACL) en suivant les stratégies précédemment développées pour P. notatum. Des analyses comparatives seront menées pour évaluer la structure du génome chez les espèces de Paspalum, et des ensembles de données transcriptomiques provenant d’inflorescences seront utilisés pour explorer l’expression génique dans la région ACL. Les travaux seront menés en collaboration avec Davide Perrone, Andrea Rubini et Fulvio Pupilli.
Photo: de gauche à droite, Fulvio Pupilli, Juan Manuel Vega & Davide Perrone

Dr. Juan Pablo A. Ortiz
IICAR, Rosario, Argentine
IRD (Montpellier, France), du 1er septembre 2025 au 30 octobre 2025
Dans le cadre des WP3 et 4, Juan Pablo A. Ortiz, de l’IICAR-CONICET (Argentine), a séjourné à l’IRD, à Montpellier (France), du 1er septembre au 30 octobre 2025. Au cours de son séjour, Juan Pablo a travaillé sur l’analyse de la transmission de l’aposporie (un élément clé de la reproduction apomictique) par les gamètes femelles. À partir des données de ségrégation générées à partir d’une plante apomictique facultative hautement sexuelle, un modèle d’hérédité de l’aposporie par les gamètes femelles est en cours d’élaboration. Le plan de travail comprenait également la détermination de la teneur en hormones (auxine, cytokinine, zéatine et acide jasmonique) dans les tissus reproducteurs des génotypes sexuels et apomictiques de Paspalum notatum. Les analyses biochimiques ont été réalisées à l’IRD par Virginie Vaissayre à l’aide de la méthode UHPLC-MS/MS. L’analyse de la concentration des hormones et de leur rôle biologique dans la reproduction apomictique sera menée en collaboration avec le Dr Olivier Leblanc.
Photo: Microscopie confocale à l’IRD, Lorena Siena (gauche) et Juan Pablo Ortiz (droite)

Dr. Maricel Podio
IICAR, Rosario, Argentine
IRD (Montpellier, France), du 1er septembre au 31 octobre 2025
Le Dr Maricel Podio, de l’IICAR (Zavalla, Argentine), a effectué un détachement de recherche à l’IRD à Montpellier, en France, du 1er septembre au 31 octobre 2025. Au cours de cette période, elle a étudié le paysage épigénétique de Paspalum notatum en utilisant la technologie Oxford Nanopore de séquençage l’ADN génomique extrait de plantes tétraploïdes sexuées et apomictiques. Afin d’initier la caractérisation de leurs épigénomes, des ovaires au stade de l’anthèse ont été sélectionnés parmi des génotypes présentant différents niveaux d’expression de l’aposporie. Le séquençage réalisé pendant le détachement permettra d’identifier et de quantifier les marques de méthylation de l’ADN (5mC), qui seront ensuite comparées aux profils de méthylation de l’ADN des feuilles afin de mieux comprendre les schémas épigénétiques associés au mode de reproduction. Toutes les expériences ont été menées en collaboration avec deux membres de l’UMR DIADE, Julien Serret et Cédric Mariac.
Photo: lab work at IRD

Sol Vega, doctorante
IICAR, Rosario, Argentine
IRD (Montpellier, France), du 1er septembre 2025 au 30 novembre 2025
María Sol Vega est accueillie à l’IRD afin de mieux caractériser le répresseur de réponse à l’auxine IAA16 chez l’espèce modèle Arabidopsis thaliana. L’orthologue de IAA16, IAA30, est régulé à la baisse dans les ovules apomictiques de Paspalum notatum par rapport aux ovules sexués. Les activités de recherche comprendront la détermination du profil d’expression spatiale de l’IAA16 dans les tissus reproducteurs à l’aide d’une construction portant le gène IAA16 fusionné avec le gène rapporteur Ruby (pIAA16:IAA16:Ruby). Des plantes doublement homozygotes seront également obtenues et étudiées, issues de croisements entre iaa16 et des lignées d’identité cellulaire de gamétogenèse et d’embryogenèse (pWOX2:CENH3, pKNU, FGR7.0, pDRN), des lignées de structuration de l’auxine (DR5, R2D2, PIN1, aux1) et des lignées mutantes pour son corépresseur TOPLESS et le gène TGS1 lié à l’apomixie. L’analyse phénotypique sera réalisée à l’aide de la cytoembryologie DIC et de l’analyse des lignées de rapporteurs spécifiques à un type cellulaire sous microscopie épifluorescente et confocale. Enfin, María Sol présentera ses travaux lors de la 5e Conférence internationale sur l’apomixie, qui se tiendra à Montpellier du 16 au 19 septembre. Le titre de sa présentation est « Auxin response repressor IAA16 defective mutants show developmental alterations in female gametophytes and embryos in Arabidopsis thaliana ».
Photo: Maria Sol Vega (gauche) & Lorena Siena (right), IRD, Montpellier (France)
Dr. José Carballo
CERZOS, Bahia Blanca, Argentine
Université de Pérouse (Italie), du 14 août au 10 octobre 2025
Dr. Juan Pablo Selva
CERZOS, Bahia Blanca, Argentine
NIAB (Royaume Uni), du 28 juillet 2025 au 27 Septembre 2025

Dr. Lorena Siena
IICAR, Rosario, Argentine
IRD (Montpellier, France), du 29 juin 2025 au 30 novembre 2025
Dans le cadre de ce détachement, Lorena Siena poursuivera la caractérisation fonctionnelle des gènes TRIMETHYLGUANOSINE SYNTHASE1 (TGS1) et QUI-GON JINN (QGJ) au cours du développement reproductif d’Arabidopsis thaliana. Les activités seront menées en collaboration avec les membres du groupe DIADE et comprendront : le séquençage d’ARN unicellulaire à l’aide de la technologie ONT à lecture longue pour analyser les différences d’expression génique entre les plantes Col et tgs1 A. thaliana et la construction de plasmides conçus pour étudier la fonction de TGS1 ; la caractérisation de l’identité cellulaire des mégaspores extra-fonctionnelles observées dans les ovules qgj, en particulier à l’aide du rapporteur pWOX2-CENH3-GFP ; et l’étude des interactions potentielles entre QGJ et TGS1 en déterminant le profil d’expression de QGJ (pQGJ-GUS) dans les ovules tgs1 et, réciproquement, celui de TGS1 (pTGS1:TGS1-GUS) dans les ovules qgj. De plus, Lorena participera à la conférence internationale Apomixis2025, qui se tiendra du 16 au 19 septembre à Montpellier, où elle a été invitée à présenter ses travaux sur la caractérisation de QUI-GON JINN lors de la reproduction femelle chez les plantes.
Photo: Lorena, Bambouseraie d’Anduze, France.

Dr. Luciana Delgado
IICAR, Rosario, Argentine
IRD (Montpellier, France), du 13 juin 2025 au 10 octobre 2025
Dans le cadre du dernier détachement du WP6 du projet MAD, en collaboration avec Daphné Autran, nous achèverons l’analyse quantitative des données relatives à la division et à la croissance cellulaires dans les primordia ovulaires de Paspalum rufum. Notre objectif est d’analyser la dynamique de la croissance et de la division cellulaires dans des contextes sexuels et apomictiques afin d’identifier les événements différentiels qui prédisent la différenciation des initiales aposporiques dans les cellules entourant la lignée germinale sexuelle chez une graminée apomictique. Cela permettra également de comprendre de manière exhaustive comment l’architecture et la forme de l’ovule influencent les processus de différenciation cellulaire pendant la formation de la lignée germinale. Nous présenterons et discuterons également nos résultats lors de la Vème Conférence internationale sur l’apomixie, qui se tiendra en septembre 2025 à Montpellier, afin de célébrer la fin de ce projet fructueux.
Photo: Luciana devant les serres de l’IRD

Dr. Michele Bellucci
IBBR, Pérouze, Italie
IICAR (Rosario, Argentine), du 29 avril 2025 au 30 mai 2025
Le 29 avril 2025, le Dr. Michele Bellucci du CNR-IBBR, Perugia, Italie, a commencé un détachement à l’IICAR jusqu’au 30 mai 2025. Il travaillera sur la transformation de Paspalum notatum par bombardement de particules, afin d’inhiber l’expression du gène PnIAA30 dans les plantes tétraploïdes sexuées et apomictiques par RNAi. Le vecteur d’inhibition a été assemblé au CNR-IBBR et contient une région du gène PnIAA30 clonée en orientation opposée et le gène bar comme marqueur sélectionnable. Nos observation ont montré que l’expression de PnIAA30 est réduite dans les ovules apomictiques par rapport aux ovules sexués. Notre hypothèse est que l’inhibition de PnIAA30 (ainsi que certains de ses paralogues) pourrait déclencher le développement d’embryons parthénogénétiques chez les plantes sexuées et/ou augmenter le taux de parthénogenèse chez les génotypes apomictiques. Toutes les expériences seront réalisées avec le Dr Carolina M. Colono et le Dr Juan Pablo A. Ortiz.
Photo: Carolina Colono, IICAR (gauche) & Michele Bellucci, IBBR (droite)
Dr. Fulvio Pupilli
IBBR, Pérouse, Italie
IICAR (Rosario, Argentine), du 5 avril 2025 au 28 mai 2025

Dr. Alessandro Camperio
University of Milan, Italy
CERZOS (Bahia Blanca, Argentine), du 17 mars 2025 au 15 avril 2025
Au cours de ce détachement, j’ai été formé à la réalisation d’analyses bioinformatiques sur un ensemble de données de séquençage d’ARN à noyau unique générées à partir d’ovaires d’Oryza sativa (échantillonnés à différents stades de développement) sous la supervision du Dr José Carballo. L’objectif était d’acquérir des connaissances en matière d’analyse transcriptomique à noyau unique et d’étudier l’expression de gènes ARF sélectionnés, identifiés chez d’autres espèces comme des gènes putatifs impliqués dans l’aposporie et la diplosporie. Dans le cadre de ce travail, j’ai cherché à générer des profils transcriptomiques à haute résolution capturant la dynamique d’expression génétique des populations cellulaires gamétophytes et sporophytes en évolution rapide pendant le développement des ovules. Ces données transcriptomiques à noyau unique seront utilisées dans un but plus large, à savoir les comparer avec d’autres ensembles de données provenant d’espèces Paspalum et Eragrostis. Ce cadre comparatif a été conçu pour mieux comprendre le paysage moléculaire découvert dans le riz, en mettant particulièrement l’accent sur la détection des gènes exprimés dans le nucelle et les tissus associés.
Photo (de gauche à droite): Viviana Echenique, Alessandro Mariani & Alessandro Camperio
Granpierro Marconi, Msc
Université de Pérouse, Italie
Université d’Alélaïde (Australie), du 5 janvier 2025 au 21 mars 2025
2024

Dr. Alessandro Mariani
Université de Pérouse, Italie
CERZOS, Bahia Blanca (Argentine), du 13 novembre 2024 au 26 mai 2025
Au cours de ce détachement au CERZOS-CONICET à Bahía Blanca, j’ai contribué au projet Mechanisms of Apomictic Development (MAD) en étudiant la régulation génétique et épigénétique de l’apomixie chez les plantes. Mes recherches ont porté sur la cartographie des loci associés aux traits apomictiques par une analyse QTL dans des populations en ségrégation, pour identifier des gènes candidats impliqués dans la diplosporie, la parthénogenèse et le développement autonome de l’endosperme. J’ai effectué une analyse bioinformatique des données de génotypage et de séquençage à haut débit, ainsi que des études de structure et association des populations, afin de découvrir les régions génomiques liées à l’apomixie. La cytométrie en flux a été utilisée pour évaluer les niveaux de ploïdie et la taille du génome, ce qui a permis de classer phénotypiquement les accessions. De plus, j’ai intégré des données sur la méthylation de l’ADN et le séquençage de l’ARN afin de détecter les différences épigénétiques entre les individus apomictiques et sexués. Ces approches intégratives ont amélioré notre compréhension des bases moléculaires de l’apomixie, conformément à l’objectif du projet qui consiste à exploiter ce trait pour améliorer les cultures.
Photo: Viviana Echenique (gauche) & Alessandro Mariani (droite) au CERZOS.
Dr. Alice Casati
Université de Milan, Italie
Unidad de Genómica Avanzada (CINVESTAV, Mexique), du 25 novembre 2024 au 15 février 2025

Giacomo Bongiorno, PhD student
Université de Pérouse, Italie
CERZOS, Bahia Blanca (Argentine), du 20 septembre 2024 au 21 novembre 2024
Au cours de ce détachement, j’ai réalisé une analyse comparative des variations de présence-absence (PAV) chez Eragrostis curvula, ce qui a conduit à l’identification de 503 gènes candidats pour l’apomixie. Plusieurs de ces gènes ont été validée par RT-PCR. J’ai également procédé à l’appel de variants (SNP et InDels) en augmentant le nombre de génotypes afin d’approfondir l’étude de la variation génomique, des modes de reproduction et des niveaux de ploïdie. L’arbre phylogénétique construit à l’aide de ces variants présente deux branches principales, distinguant les génotypes à ploïdie elevée des génotypes diploïdes et tétraploïdes. Cette analyse a permis de mieux comprendre les relations génétiques entre les génotypes de différents niveaux de ploïdie, mettant en lumière de nouveaux scénarios liés à l’évolution du génome chez E. curvula.
Photo: Giacomo Bongiorno (gauche) et José Carballo (droite)

Ing. Caroline Michaud
IRD, Montpellier, France
IICAR, Rosario (Argentine), du 12 novembre 2024 au 13 décembre 2024
Au cours de cette misison, j’ai contribué aux travaux de Luciana Delgado et Daphné Autran sur Paspalum rufum en explorant les liens entre l’architecture du primordium de l’ovule et la plasticité du destin cellulaire menant à l’aposporie. Pour déterminer les schémas de prolifération cellulaire au cours du développement de l’ovule chez Paspalum rufum sexué et apomictique aux stades pré-méiotiques, nous avons réalisé des expériences pour marquer les cellules en phase S du cycle cellulaire à l’aide du kit Click-iT™ EdU. Les ovaires ont été disséqués à quatre stades précoces, traités pour l’incorporation et la détection de l’EdU, puis imagés à l’aide d’un microscope confocal ZEISS 880 à l’Institut de biologie moléculaire et cellulaire (IBR-CONICET) de Rosario. Ces images seront segmentées avec MorphographX (MGX) pour quantifier les taux de prolifération différentiels entre les génotypes. J’ai également collaboré à un projet d’Olivier Leblanc et Lorena Siena, visant à construire des plasmides pour l’analyse fonctionnelle du gène TGS1 chez Arabidopsis thaliana, dont la caractérisation est prévue à Montpellier en 2025.
Photos. Gauche haut : ma carte d’enseignante de l’UNR! ; Gauche bas : le microscope confocal de l’IBR, et; Droite: un diagramme des stades disséqués de Paspalum rufum.

Dr. Olivier Leblanc
IRD, Montpellier, France
IICAR, Rosario (Argentine), du 12 novembre 2024 au 13 décembre 2024
Photo: traverser la Pampa de Rosario à Bahia Blanca…
Dr. Tamunonengiye-ofori Lawson
Cambridge, Royaume Uni, NIAB
CERZOS (Argentine), du 31 octobre 2024 au 15 décembre 2024

Dr. Daphné Autran
IRD, Montpellier, France
IICAR, Rosario (Argentine), du 28 octobre 2024 au 31 décembre 2024
Au cours de ce détachement, dans le cadre du WP6 du projet MAD, Daphné Autran (IRD) a collaboré avec Luciana Delgado (IICAR-CONICET), avec l’aide de Caroline Michaud (IRD), pour décrypter la morphogenèse de l’ovule chez l’espèce aposporique Paspalum rufum. Pour déterminer quels sont les premiers événements cellulaires associés à un programme de développement aposporique facultatif, nous avons profité de la période de floraison de l’unique population de plantes de P. rufum présentant des phenotypes reproducteurs contrastés et disponible à l’IICAR, Zavalla, Rosario, Argentine. Ici, le processus de prolifération cellulaire a été spécifiquement abordé, en caractérisant le nombre et la localisation des cellules en division pour des ovules vivants collectés à différents stades de développement. Après fixation, nettoyage et coloration, les échantillons ont été imagés par microscopie confocale à fluorescence (disponible à l’IBR, Instituto de Biologia de Rosario). L’ensemble du jeu d’images 3D générées sera ensuite analysé dans le but de compléter les informations recueillies dans l’atlas de croissance cellulaire 3D des ovules de P. rufum produit lors de précédents détachements.
Image: Luciana Delgado (gauche) & Luciana Delgado et Daphné Autran (colonne droite)

Dr. Silvina Pessino
IICAR, Rosario, Argentine
Université de Milan (Italie), du 2 octobre 2024 au 27 novembre 2024
Silvina Pessino (IICAR-CONICET, Argentine) a été détachée à l’UMIL, Italie, du 2 octobre au 27 novembre 2024, pour étudier le rôle de plusieurs AUXIN RESPONSE FACTORS (ARF) et de leurs régulateurs miRNA respectifs dans la spécification de la lignée germinale femelle chez Arabidopsis thaliana, en collaboration avec les Dr. Lucia Colombo et Marta Mendes. En particulier, au cours de ce détachement, elle a participé aux étapes finales de la publication de l’article Pessino SC et al. (2024) AUXIN RESPONSIVE FACTOR 10 insensitive to miR160 regulation induces apospory-like phenotypes in Arabidopsis, iScience; préparé plusieurs livrables du projet MAD, et analysé les données McSeED et RNA-seq indiquant le contrôle de l’expression de l’ARF médié par le système RdDM.
Photo: marta mendes (gauche) & Silvina Pessinio (droite)

Dr. Lorena Siena
IICAR, Rosario, Argentine
Université de Padoue (Italie), du 1er octobre 2024 au 1er novembre 2024
Au cours de ce détachement, Lorena Siena analysera le profil d’expression du gène BUD13 au cours du développement reproducteur des plantes apomictiques de Paspalum notatum et d’Hypericum perforatum. Lors d’un précédent détachement MAD, il a été constaté que BUD13 est exprimé de manière différentielle entre les génotypes apomictiques et sexué chez les deux espèces. Il est intéressant de noter que les mutants BUD13 d’Arabidopsis présentent des défauts au cours du développement embryonnaire précoce. Les activités de recherche comprendront la conception d’amorces PCR pour amplifier les séquences génomiques et d’ARNm, des analyses d’expression par qRT-PCR et l’analyse d’images obtenues à l’IICAR-CONICET par hybridation in situ sur d’ovaires de P. notatum. De plus, les principaux résultats de l’étude seront discutés dans le but de preparer une publicatoion. Ce travail est réalisé en collaboration avec Samela Draga dans le cadre de sa thèse de doctorat au DAFNAE. Les informations obtenues contribueront au WP4 et pourraient fournir un nouveau candidat pour la parthénogenèse.
Image: Collection de plantes d’Hypericum perforatum en serre

Dr. Juan Pablo A. Ortiz
IICAR, Rosario, Argentine
Université de Padoue (Italie), du 1er octobre 2024 au 1er novembre 2024
Ce nouveau détachement de Juan Pablo A. Ortiz a pour objectif d’analyser des séquences liées à l’apomixie, avec un accent particulier sur la découverte de gènes associés à la parthénogenèse. Ce travail comprendra l’analyse de bases de données transcriptomiques de génotypes sexués et apomictiques afin d’identifier les gènes qui participent aux cascades de réactions hormonales, aux différentes étapes de traitement de l’ARN et à la signalisation cellulaire. L’analyse inclura également les ressources génomiques disponibles sur les locus de contrôle de l’apomixie disponibles pour différentes plantes apomictiques. Ce travail sera réalisé en collaboration avec le Dr Giovanni Gabelli du groupe DAFNAE. Les résultats obtenus contribueront aux WP3 et WP4 et pourraient fournir de nouveaux candidats pour l’induction de la parthénogenèse chez les espèces apomictiques naturelles.
Photo: Partage du maté… de gauche à droite: Juan Pablo Ortiz, Lorena Siena & Samela Draga

Dr. Lorena Siena
IICAR, Rosario, Argentine
IBBR (Italie), du 30 août 2024 au 29 septembre 2024
Pendant sa visite au laboratoire du Dr. Fulvio Pupilli, Lorena séquencera des plantes tétraploïdes sexuelles et apomictiques de Paspalum simplex afin d’améliorer les assemblages actuels de génomes tétraploïdes. Les informations produites contribueront au WP3 et seront utilisées pour étudier la structure et l’évolution du génome et pour déchiffrer le contenu génique du locus d’apomixie. Ce travail sera réalisé en collaboration avec le Dr Andrea Rubini et les BC Davide Perrone de l’IBBR et le Dr Juan Pablo A Ortiz de l’IICAR. Lorena mettra également en place des tests d’hybridation in situ pour caractériser le profil d’expression du gène candidat ARF5 de P. simplex dans les ovaires apomictiques et sexuels. Elle participera à la conception et à l’étiquetage de la sonde, ainsi qu’au montage en paraffine des ovaires. Le sectionnement et l’hybridation seront effectués à l’IICAR, CONICET-UNR. Ces expériences seront réalisées en collaboration avec Dr Eugenia Cáceres de l’IBBR.
Image: Lorena Siena (droite) & Davide Perrone (gauche)

Dr. Juan Pablo A. Ortiz
Rosario, Argentina, IICAR
IBBR (Italie), du 30 août 2024 au 29 septembre 2024
L’objectif de ce détachement est d’approfondir la caractérisation des génomes sexués et apomictiques chez Paspalum simplex. Le plan de travail comprend : extractions d’ADN, préparation de bibliothèques et séquençage ONT de plantes tétraploïdes. Ces activités contribueront au WP3, qui vise à analyser la structure et l’évolution des génomes apomictiques et à élucider le contenu en gènes et la fonction du locus d’apomixie. Les autres membres de l’équipe qui participeront à ce travail sont : Dr Lorena Siena (IICAR), Dr Andrea Rubini (IBBR) et BSc Davide Perrone (IBBR). Juan Pablo travaillera également à la construction d’un vecteur RNAi conçu pour réduire l’expression de ORC3, un gène candidat identifié par le groupe du Dr Pupilli comme régulateur du développement de l’albumen dans les graines apomictiques. Cette construction sera utilisée pour générer des plantes de maïs transgéniques afin de valider sa fonction dans le contrôle de l’excès génomique maternel dans l’albumen d’une plante sexuée cultivée. Cette partie du travail sera réalisée en collaboration avec Dra Francesca De Marchis et Dr Michelle Bellucci, de l’IBBR.
Image: Juan Pablo A. Ortiz (gauche) & Fulvio Pupilli (droite)

Ing. David Lopez Villegas
CERZOS, Bahia Blanca, Argentine
IRD (France), du 20 juin 2024 au 26 août 2024
David Lopez Villegas (Bahia Blanca, Argentine) a effectué un détachement à l’IRD à Montpellier, en France, du 24 juin au 23 août 2024. Au cours de ce détachement, il a acquis les compétences et les connaissances nécessaires pour mener à bien la valorisation technologique en collaboration avec le Bureau de transfert de technologie de l’IRD, en mettant l’accent sur l’apomixie et ses applications potentielles dans l’agriculture. Il a procédé à une analyse systématique des brevets liés à l’apomixie et aux technologies associées, fournissant ainsi des informations précieuses sur les tendances technologiques actuelles, les stratégies en matière de propriété intellectuelle et les principales institutions et entreprises actives dans ce domaine.
Photo: David Lopez, IRD, Montpellier, France.

Dr. Andrés Bellido
CERZOS, Bahia Blanca, Argentine
IRD (France), du 31 mai 2024 au 29 juin 2024
Des plantes transgéniques d’Arabidopsis thaliana ont été générées en exprimant des gènes liés à l’apomixie et identifiés chez Eragrostis curvula. Celles-ci expriment plusieurs phénotypes liés à l’apomixie lors de la spécification de la CMM et au cours du development du sac embryonnaire, ainsi que des signes de perte de méthylation de l’ADN. L’objectif de ce travail est de caractériser les marques d’histones les plus pertinentes au cours du développement de l’ovule dans ces lignées transgéniques d’Arabidopsis en utilisant des techniques d’immunomarquage et de microscopie confocale.

Dr. Viviana Echenique
CERZOS, Bahia Blanca, Argentine
Université de Pérouse (Italie), du 30 mai 2024 au 2 août 2024
L’objectif de ce détachement à l’UNIPG est de poursuivre le travail avec le groupe du Dr. Albertini pour identifier la région génomique impliquée dans l’apomixie chez Eragrostis curvula et pour caractériser les gènes présents. Pour cela, nous analyserons les données précédemment obtenues à partir du pangénome d’E. curvula, qui consiste en un séquençage Illumina de 10 génotypes présentant des génomes similaires mais avec des différences pour les modes de reproduction (apomictique et sexué) et les niveaux de ploïdie (2x, 4x, 6x, 7x et 8x). Les données seront utilisées pour étudier les régions génomiques impliquées dans l’apomixie et leur synténie avec leurs régions homologues sexuées par une approche de cartographie afin d’obtenir le premier assemblage du pangénome d’E. curvula.

Dr. Maria Cielo Pasten
CERZOS, Bahia Blanca, Argentine
Université de Pérouse (Italie), du 30 mai 2024 au 2 août 2024
L’objectif de ce détachement était d’initier la caractérisation de lignées mutantes SALK chez Arabidopsis thaliana pour les gènes CHC2 (Clathrin heavy chain 2) et NFD3 (Nuclear fusion defective 3), tous deux précédemment identifiés comme exprimés de manière différentielle dans les génotypes apomictiques et sexués d’Eragrostis curvula. Le génotypage avec des amorces spécifiques a été réalisé et plusieurs des lignées hétérozygotes qui ont été identifiées sont maintenant étudiées. Nous avons également obtenu des plantes mutées par CRISPR-Cas9 pour le gène SPL7 (Squamosa promoter binding like 7). Celles-ci sont en cours de caractérisation. De plus, des hybridations in situ (ISH) ont été réalisées afin de determiner le profil d’expression du gène SPL7 dans les tissus reproducteurs d’individus diploïdes et tétraploïdes chez A. thaliana (ecotype Col0).

Domenico Loperfido, MSc
Université de Milan, Italie
CERZOS (Bahia Blanca, Argentine), du 11 mars 2024 au 8 juin 2024
Au cours des trois mois (du 11 mai au 8 juin 2024) qu’il a passés au CERZOS, Domenico Loperfido a travaillé dans le laboratoire du professeur Echenique sous la supervision de José Carballo et de Cristian Gallo du CERZOS et celle du professeur Ezquer de l’Université de Milan. L’objectif du détachement était d’étudier les gènes candidats impliqués dans la croissance et le développement des graines par des études d’association visant à comprendre le rôle que ces gènes peuvent avoir dans l’hérédité des processus apomictiques. Les génomes de différentes accessions d’Arabidopsis thaliana ont été utilisés ; en utilisant des outils bioinformatiques, par exemple bcftools, vcftools, R, GAPIT, et d’autres, des SNP significatifs dans les gènes phénotypiquement associés à la croissance et au développement des composants de la graine (embryon, endosperme, et tégument) contrôlant la vigueur hybride dans les plantes sexuées ont été identifiés.

Dr. Diego Zappacosta
CERZOS, Bahia Blanca, Argentine
Université de Pérouse (Italie), du 5 mars 2024 au 5 mai 2024
L’objectif principal de ce détachement est de poursuivre la caractérisation d’une population en segregation pour l’apomixie chez Eragrostis curvula pour générer une carte de liaison et sélectioner des individus présentants des marqueurs moléculaires similaires mais des modes de reproduction différents (apomictique et sexué) pour de futures analyses transcriptomiques. Pendant ces deux mois, en collaboration avec le groupe de l’UNIPG, je vais phénotyper et caractériser moléculairement des individus de la population de cartographie. Notre objectif est de créer une nouvelle carte consensus pour E. curvula, d’identifier des marqueurs moléculaires liés à l’apoméiose et, finalement, de découvrir des gènes qui pourraient jouer un rôle dans le contrôle de la reproduction apomictique. De plus, je vais aussi contribuer à la production d’un calendrier visant à associer les paramètres morphologiques floraux aux stades de développement de la mégasporogenèse et de la mégagamétogenèse pour différents génotypes apomictiques et sexués chez E. curvula (Task 3.3).
Lucia Giordano, doctorante
Université de Pérouse, Italie
Université d’Adélaïde (Australie), du 18 janvier 2024 au 2 juin 2024
2023
Samela Draga, doctorante
Université de Padoue, Italie
IICAR (Rosario, Argentine), du 3 novembre 2023 au 19 janvier 2024
La visite de Samela Draga au laboratoire du Prof. Pessino du 3 novembre 2023 au 18 janvier 2024 apour objectif général de poursuivre le projet initié en mars-juillet 2023 à l’IICAR-CONICET-UNR qui vise à comprendre le rôle du facteur d’épissage BUD13 (un interacteur de TGS1) chez les plantes de Paspalum apomictiques et sexuées. BUD13 présente une expression différentielle dans les fleurs des plantes sexuées et apomictiques chez au moins deux espèces aposporiques, Paspalum notatum et Hypericum perforatum. Il est intéressant de noter que les mutants bud13 d’Arabidopsis sont associés à des défauts du développement embryonnaire précoce. Au cours de sa visite, Samela produira une sonde BUD13 marquée à la digoxigénine, préparera des coupes d’ovaires inclus dans des blocs de paraffine, et effectuera l’analyse ISH. Les observations permettront de déterminer les profils d’expression spatiale de BUD13 dans les organes reproducteurs des génotypes sexuels et apomictiques de Paspalum notatum.
Dr. Giovanni Gabelli
Université de Pérouse, Italie
IICAR (Rosario, Argentine), du 15 novembre 2023 au 15 décembre 2023

Dr. Fulvio Pupilli
IBBR, Pérouse, Italie
IICAR (Rosario, Argentine), du 8 novembre 2023 au 9 décembre 2024
Au cours de son premier détachement à Rosario du 9 novembre au 14 décembre 2023, Fulvio Pupilli a discuté avec Juan Pablo Ortiz, son équipe et Olivier Leblanc les récentes avancées concernant le séquençage Nanopore et l’assemblage de génomes apomictique et sexué chez Paspalum simplex. De plus, les progrès réalisés dans la caractérisation du gène PsARF5 qui est exprimé de manière différentielle entre les tissus du nucelle disséqué par LCM de génotypes apomictiques et sexuels ont été discutés avec JP Ortiz ainsi que les interactions possibles avec PnIAA30, un gène en cours de caractérisation fonctionnelle à l’IICAR. Une série d’expériences d’hybridation in situ pour PsARF5 a été programmée pour le prochain détachement. Enfin, il a préparé un exposé sur le rôle du gène PsORC3 dans le développement des l’albumens présentant un excès génomique maternel chez P. simplex et un hommage au professeur Camilo Luis Quarin pour le IVe congrès Apomixis, qui se tiendra du 3 au 7 décembre 2023 à Rosario, en Argentine.

Giacomo Bongiorno, doctorant
Université d Pérouse, Italie
CERZOS (Bahia Blanca, Argentine), du 18 mai 2023 au 9 décembre 2023
Pendant ce détachement au CERZOS, je me suis principalement concentré sur la graminée diplosporique Eragrostis curvula dans le but de construire un pan-génome complet. L’utilisation du cultivar Victoria comme génome de référence a fourni une base solide pour étudier la variation génomique, les relations phylogénétiques et l’impact de la ploïdie sur l’évolution du génome et les modes de reproduction. Cette étude pan-génomique contribue de manière significative à notre compréhension de la diversité génomique d’E. curvula, comme ressource d’intérêt pour des investigations ciblées sur les mécanismes moléculaires régissant ses modes de reproduction et ses relations avec la ploïdie.
Photo (de gauche à droite): Juan Pablo Selva, Diego Zappacosta, Juan Manuel Rodrigo, Nengi Lawson, Viviana Echenique, Alejandra Diaz & Giacomo Bongiorno

Dr. Daphné Autran
IRD, Montpellier, France
IICAR (Rosario, Argentine), du 22 octobre 2023 au 17 décembre 2023
Au cours de ce détachement, nous poursuivons le travail sur Paspalum rufum avec Luciana Delgado. Pour comprendre les interrelations entre l’architecture des primordia de l’ovule et la plasticité du destin cellulaire menant à l’aposporie, nous effectuons une analyse morphométrique cellulaire en 3D des génotypes de P. rufum montrant une expressivité différentielle de l’aposporie. La première étape consiste à construire un atlas 3D de référence du génotype sexué. Après le travail de segmentation, d’étiquetage et d’exportation des données effectué lors des précédents détachements à l’aide du logiciel MorphographX (MGX), nous avons mis en place une analyse quantitative sur un groupe pilote de 11 ovules à des stades successifs couvrant la fenêtre de développement qui nous intéresse. En plus de la quantification du nombre et de la taille des cellules, différents descripteurs de forme, au niveau des cellules et des organes, ont été sélectionnés et calculés à partir des caractéristiques extraites par MGX. Comme les données pilotes comprennent déjà des milliers de cellules à analyser, des scripts personnalisés en R ont été écrits pour compiler, visualiser et analyser statistiquement les données. En outre, les ovules de P. rufum au stade de la méiose ont été disséqués, fixés et nettoyés, afin d’étendre la fenêtre de développement couverte et de maximiser la probabilité de détection de la croissance cellulaire différentielle entre les génotypes sexués et aposporiques.
Paspalum rufum, Luciana Delgado & Daphné Autran

Dr. Olivier Leblanc
IRD, Montpellier, France
IICAR (Rosario, Argentine), du 15 octobre 2023 au 13 décembre 2023
Cette deuxième visite à l’IICAR s’inscrit dans un détachement consacré à la génomique de l’apomixie (WP3) et à la caractérisation fonctionnelle de gènes candidats de Paspalum pour l’apomixie en utilisant des mutants d’Arabidopsis (WP4). En ce qui concerne les approches génomiques, une premier objectif est de finaliser la rédaction d’un premier article rapportant l’assemblage et l’annotation détaillée d’un génome diploïde de référence de P. notatum. Par ailleurs, nous concentrerons nos analyses sur la compréhension des variations entre le génome des plantes diploïdes sexuées et celui des plantes tétraploïdes apomictiques en utilisant des approches de génomique comparative et de k-mers. En ce qui concerne la caractérisation fonctionnelle des mutants d’Arabidopsis, les effets des mutations tgs1 sur l’épissage de l’ARN seront analysées à l’aide d’analyses transcriptionnelles comparatives et d’outils cytologiques (rapporteurs fluorescents en cours de construction). Par ailleurs, je participe à l’organisation du deuxième atelier MAD sur le séquençage et la bioinformatique de l’ONT (Rosario, 20-25 novembre) et de la conférence Apomixis2023 (Rosario, 3-7 décembre).
Photo: visite de vieilles connaissances à Cayasta (Santa Fe, Argentine), berceau de la famille Paspalum. De gauche à droite : O Leblanc, JPA Ortiz, & L Siena

Davide Perrone
Université de Milan, Italie
IICAR (Rosario, Argentine), du 12 septembre 2023 au 13 décembre 2023
Davide Perrone est en visite à l’IICAR-CONICET pour poursuivre les expériences initiées lors d’un précédent détachement, concernant la transformation de plantes apomictiques et sexuées de Paspalum notatum avec des rapporteurs fluorescents d’auxine et de cytokinine. Le but de ces expériences est de comparer la distribution des hormones dans les ovules de plantes sexuées et apomictiques. De plus, Davide initiera des expériences d’hybridation in situ visant à comparer l’expression du gene AUXIN RESPONSE FACTOR 10 (ARF10) dans les ovules des biotypes sexués et apomictiques de Paspalum notatum. Enfin, en collaboration avec les scientifiques de l’IICAR, il conduira des analyses miR-seq en utilisant les données générées précédemment à Milan.
Photo (de gauche à droite): Augustus Villalba (IBONE), Silvina Pessino (IICAR), Maricel Podio (IICAR) & Davidse Perrone (Univ. Milan)
Dr. Cristian Gallo
Bahia Blanca, Argentina, CERZOS
Université de Pérouse (Italie), du 1er septembre 2023 au 30 octobre 2023

Dr. Francesca De Marchis
Pérouse, Italie, CNR-IBBR
IICAR (Rosario, Argentine), du 11 août 2023 au 12 septembre 2023
Francesca De Marchis est une chercheuse du Conseil national de la recherche, CNR- IBBR – Perugia. Pendant son séjour, elle va collaborer avec Juan Pablo Ortiz (IICAR) et son expertise en biotechnologie végétale sera mise à profit pour concevoir un vecteur de transformation pour Paspalum notatum. Certains gènes candidats pour le déclenchement de la parthénogenèse, et identifiés par le groupe de recherche de l’IICAR, seront sélectionnés pour la transformation de plantes par la technologie biolistique dans le but de modifier leur profil d’expression (expression ectopique ou repression).

Dr. Alejendra Diaz
Bahia Blanca, Argentine, CERZOS
NIAB (Royaume Uni), du 1er aout 2023 au 2 octobre 2023
Notre groupe de recherche CERZOS a engagé une collaboration depuis plusieurs années avec le groupe du Dr. Caccamo au NIAB visant à l’identification et la caractérisation d’une région génomique impliquée dans l’apomixie chez Eragrostis curvula (Schrad.) Nees, et de ses gènes. Certains d’entre eux, identifiés précédemment par notre groupe de recherche comme impliqués dans l’apomixie et pour lesquels aucunne annotation chez d’autres espèces n’est disponible, sont en cours de caractérisation fonctionnelle. L’expression de ces candidats sous des promoteurs constitutifs dans la plante modèle qu’est le riz est l’une des approches utilisées pour leur caractérisation. L’objectif de ce détachement au NIAB était de faire progresser la caractérisation de l’un de nos gènes candidats, principalement pour déterminer si sa surexpression entraînait un défaut dans le développement du sac embryonnaire. Pour ce faire, j’ai participé au criblage de tissus reproducteurs provenant d’épillets de lignées de riz transgéniques indépendantes générées au NIAB et surexprimant le gène d’intérêt (WP4). Nous avons utilisé la microscopie à contraste d’interférence différentiel (DIC) pour observer les tissus reproducteurs nettoyés avec une solution de salicylate de méthyle, car cette technique permet des observations à grande échelle sur la méiose et le développement du sac embryonnaire dans des ovules intacts. Ainsi, la collaboration entre le NIAB et le CERZOS nous permettra de caractériser plus rapidement les gènes candidats à l’apomixie identifiés chez Eragrostis.

Dr. Viviana Echenique
Bahia Blanca, Argentine, CERZOS
NIAB (Royaume Uni), du 1er août 2023 au 2 octobre 2023
L’objectif de mon détachement au NIAB est de poursuivre le travail initié avec le groupe du Dr. Caccamo visant l’identification et la caractérisation d’une région génomique et de gènes impliqués dans l’apomixie. Différentes stratégies et de nouvelles experiences ont été discutées. Le travail en cours consiste à séquencer et à assembler le génome du cultivar apomictique Don Walter (Eragrostis curvula). Ce génome a été séquencé au NIAB à l’aide de la plateforme Oxford nanopore avec le protocole de modèle végétal de très haute précision et l’un des étudiants du CERZOS travaille ici à son assemblage. Nous sommes également impliqués dans un projet de transcriptomique utilisant des données RNA-seq obtenues à partir de tissus de pistil de plantes apomictiques et sexuées à trois stades de développement différents. Nous recherchons des gènes différentiellement exprimés entre des plantes ayant des modes de reproduction différents et des stades de développement différents.
Photo: Viviana devant The Eagle à Cambridge (Angleterre)

Sol Vega, doctorante
Rosario, Argentina, IICAR
IRD (France), du 20 juillet 2023 au 20 septembre 2023
Ce détachement de María Sol à l’IRD à pour objectif de poursuivre la caractérisation fonctionnelle du gène IAA16 chez Arabidopsis thaliana, dont l’orthologue (IAA30) a été associé à l’apomixie chez Paspalum notatum. Les activités seront menées dans le laboratoire du Dr. Olivier Leblanc et viseront l’obtention de plantes double-t homozygotes issues de croisements entre le mutant iaa16 et une lignée portant le rapporteur gamétophytique pWOX2-CENH3-GFP ; des plantes mutantes tgs1 (liées à des altérations du développement des gamétophytes), et des plantes mutantes topless (réponse à l’auxine au cours de l’embryogenèse). Des analyses phénotypiques du développement reproducteur des matériels obtenus seront effectuées en utilisant la microscopie à fluorescence ou DIC pendant le détachement et ensuite à l’IICAR. Par ailleurs, l’étude de mutants pour d’autres gènes du clade IAA16 (iaa14 et iaa17) ainsi que pour IAA16 a été initiée.

Dr. Lorena Siena
Rosario, Argentina, IICAR
IRD (France), du 22 juin 2023 au 16 septembre 2023
Dans le cadre de ce détachement, Lorena Siena, de l’IICAR, sera accueillie à l’IRD pendant trois mois. L’objectif principal est de faire progresser la caractérisation fonctionnelle des gènes TGS1 et QGJ au cours du développement reproducteur chez Arabidopsis thaliana. Les activités seront menées en collaboration avec le groupe de l’IRD e viseront, tout d’abord, à explorer le potentiel pour la formation d’embryons parthénogénétiques dans les lignées mutantes tgs1 en utilisant des rapporteurs fluorescents spécifiques de l’embryon et, transmis maternellement ou paternellement. En outre, l’identité cellulaire des mégaspores, supplémentaires et fonctionnelles, observées dans les ovules qgj sera caractérisée de manière approfondie, par exemple en utilisant le rapporteur pWOX2-CENH3-GFP. De plus, nous initierons des expériences visant à identifier les interactions potentielles entre QGJ et TGS1 en déterminant le profil d’expression de QGJ (pQGJ-GUS) dans les ovules tgs1 et, réciproquement, celui de TGS1 (pTGS1:TGS1-GUS) dans les ovules qgj.
Image: Aqueduc de Montpellier (@ Lorena Siena, IICAR)

Dr. Jose Carballo
Bahia Blanca, Argentina, CERZOS
NIAB (Royaume Uni), du 14 juin 2023 au 14 septembre 2023
L’objectif de ce détachement au NIAB est de poursuivre le séquençage et l’assemblage du génome apomictique d’Eragrostis curvula. Le génome du génotype Don Walter a été séquencé au NIAB en utilisant la plateforme Oxford nanopore avec le protocole du modèle de plante de très haute précision. Les lectures ont été assemblées à l’aide du logiciel Flye et échafaudées avec la technologie Omni-C afin d’obtenir un assemblage à l’échelle du chromosome. Le génome assemblé a ensuite été annoté pour obtenir les sous-génomes A et B de l’allotetraploïde Don Walter. De plus, pendant cette visite à Cambridge, les données RNA-seq, obtenues à partir de pistils de plantes apomictiques et sexuées à trois stades de développement différents, ont été analysées pour identifier les gènes différentiellement exprimés. Ce détachement poursuit les efforts de collaboration entre le NIAB et le CERZOS-CONICET afin de caractériser les bases moléculaires de l’apomixie par une approche bioinformatique.

Dr. Tamunonengiye-ofori Lawson
Cambridge, UK, NIAB
CERZOS (Argentine), du 20 mai 2023 au 19 juillet 2023
L’objectif de ce séjour au CERZOS était d’approfondir mes connaissances, mes compétences et mon expérience pratique dans le domaine de l’hybridation in-situ et de la microscopie DIC chez les plantes.
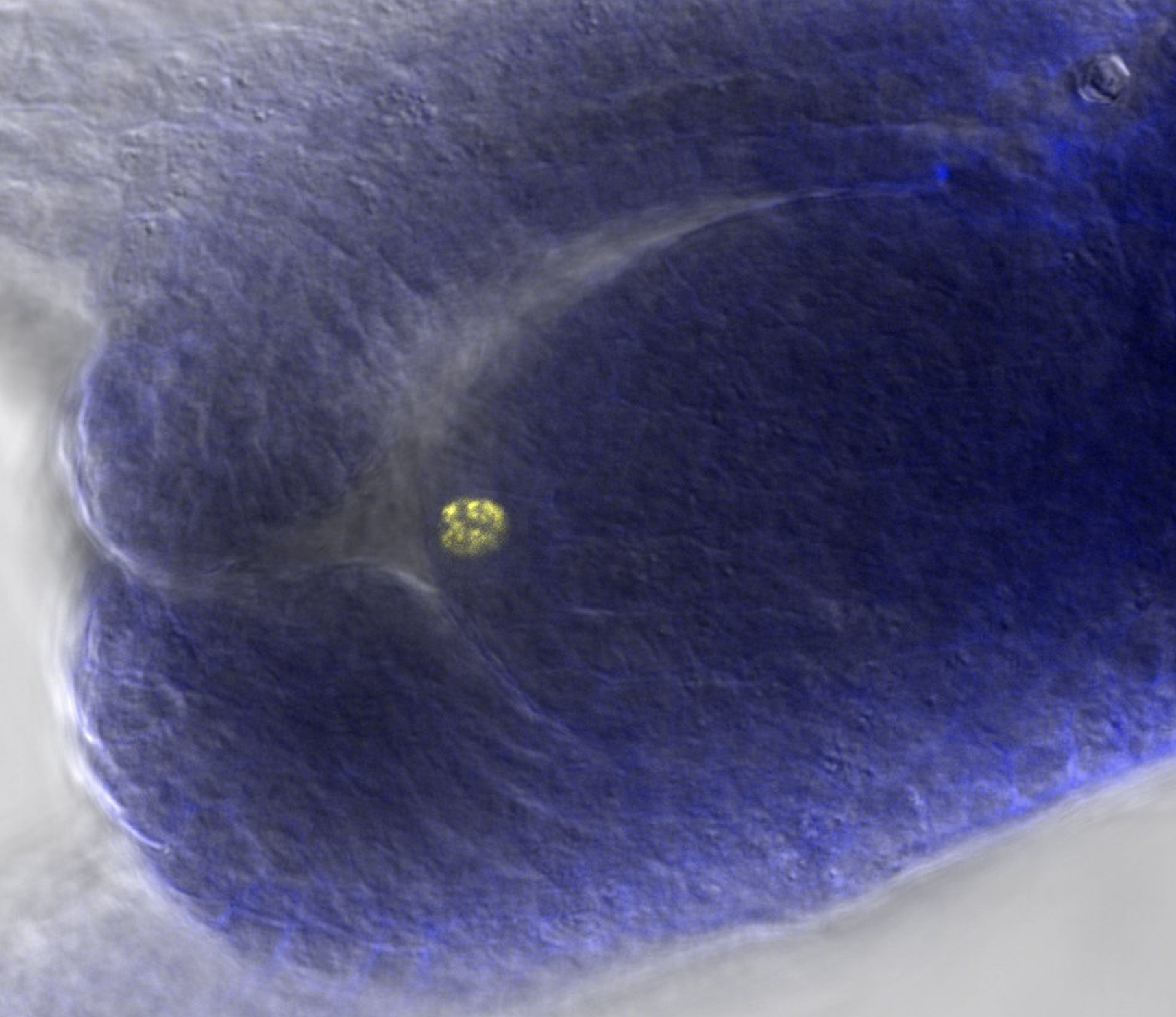
Dr. Luciana Delgado
Rosario, Argentine, IICAR
IRD (France), du 20 mai 2023 au 19 août 2023
Cette mission s’inscrit dans la continuité des travaux initiés en 2022 avec Daphné Autran visant à l’obtention d’un atlas 3D du développement précoce de l’ovule chez Paspalum rufum. Nous réaliserons la segmentation et la classification des cellules des primordia d’ovules à l’aide d’images haute résolution obtenues précédemment et un workflow partiellement automatisé préalablement établi utilisant le logiciel libre MorphoGraphX. De plus, nous testerons différents protocoles d’incorporation et détection d’un analogue de la thymidine (EdU) pour quantifier les divisions cellulaires au cours du développement de l’ovule chez différentes graminées, dont deux espèces modèles sexuées (le riz et le maïs) et des plantes apomictiques de Paspalum, Ces protocoles doivent être optimisés pour les ovules de graminées de morphologies diverses. Les informations quantitatives ainsi collectées à partir de l’atlas 3D et des divisions cellulaires pour des ovules de plantes sexuées et apomictiques améliorera notre compréhension de la transition entre sexualité et apomixie.
Photo : Cellule en interphase dans un jeune ovule de Paspalum rufum présentant un signal fluorescent de l’EdU incorporé à l’ADN (@ Luciana Delgado, IICAR).

Samela Draga, doctorante
Université de Padoue, Italie
IICAR (Rosario, Argentine), du 16 mai 2023 au 8 août 2023
Le travail de Samela à l’IICAR a pour objectif principal de contribuer à l’élucidation du rôle dans la transition reproduction sexuée-apomixie de la protéine BUD13, requise pour l’épissage alternatif. En particulier, elle caractérisera BUD13 dans les bases de données transcriptomiques et génomiques de Paspalum notatum et générera des sondes pour leur détection par hybridation in-situ des ARNs. De plus, elle classera les ovules de plantes de Paspalum notatum sexuées et apomictiques en fonction des stades de développement en utilisant la cytoembryologie DIC, les déshydratera dans une série éthanol/xylol et les inclura dans de la paraffine. Lors d’une deuxième visite (octobre-décembre), elle préparera des sections d’ovule pour hybridation avec des sondes d’ARN sens et antisens marquées à la digoxigénine et détectées par réaction colorimétrique (anticorps DIG conjugué à la phosphase alcaline, substrats NBT/BCIP). Nous espérons obtenir des informations sur l’expression spatiale de ces gènes candidats pour des génotypes apomictiques et sexués.
Samela Draga (droite) & Silivina Pessino (gauche)

Nicola Babolin, doctorant
Université de Milan, Italie
Université d’Adélaide (Australie), du 10 avril 2023 au 13 octobre 2023
Mon détachement à l’Université d’Adélaïde (Australie) dans le laboratoire du Prof. Matthew Tucker a pour objectif de mieux caractériser le réseau moléculaire établi entre les deux gènes MADS-box, STK et ABS, au cours de la fécondation et du développement de la graine chez Arabidopsis thaliana.
Je serai aussi impliqué dans les projets menés par l’équipe du prof. Tucker sur l’orge concernant le développement de l’ovule et de la graine, fortement interconnectés avec mon projet sur Arabidopsis.
De gauche à droite: Nicola Babolin, Matthew Tucker, Rosanna Petrella

Dr. Rosanna Petrella
Université de Milan, Italie
Université d’Adélaide (Australie), du 10 avril 2023 au 13 octobre 2023
Pendant mon détachement à l’Université d’Adélaïde (laboratoire de Matthew Tucker), mon travail se concentrera sur l’élucidation des mécanismes moléculaires déterminant la progression de la lignée germinale femelle chez Arabidopsis thaliana. En particulier, j’essaierai de caractériser les principaux acteurs impliqués dans la dégénérescence des spores et dans la sélection et la différenciation fonctionnelles des mégaspores à la fin de la mégasporogenèse dans l’ovule. Je déterminerai la contribution de l’interaction entre les phytohormones en essayant de sauver les mutants défectueux dans ces processus clés en utilisant des régions régulatrices spécifiques actives dans différents domaines de l’ovule pour piloter spécifiquement l’expression des gènes candidats.
De gauche à droite: Rosanna Petrella, Skippy le Kangourou, Nicola Babolin

Mattia Spano, doctorant
Université de Milan, Italie
LANGEBIO (Mexique) du 1er avril 2023 au 20 juillet 2023
L’objectif de ce détachement est d’étudier les mécanismes moléculaires qui déterminent la lignée germinale femelle. En particulier, j’étudierai les contributions des phytohormones, l’auxine et les cytokinines, dans la différenciation de la CMM. Les espèces étudiées sont Arabidopsis thaliana et Oryza sativa var. Kitaake. Dans ce cadre, j’effectuerai des tests d’immunolocalisation dans différents contextes pour localiser les phytohormones pendant le développement de l’ovule en utilisant le mutant spl/nzz et le type sauvage d’Arabidopsis et le type sauvage d’Oryza sativa.

Dr. Juan Pablo A. Ortiz
Rosario, Argentine, IICAR
IRD (France), du 7 mars 2023 au 3 avril 2023
Un nouveau séjour à l’IRD du Dr. Juan Pablo A. Ortiz de l’IICAR, effectué dans le cadre d’un détachement MAD pour la caractérisation des génomes apomictiques (WP3), a débuté le 7 mars 2023. Le plan de travail comprend l’extraction d’ADN de plusieurs échantillons de Paspalum spp, destiné au séquençage Illumina pour réaliser des analyses structurelles du génome. La visite est également consacrée à la préparation d’un nouveau manuscrit décrivant le séquençage, l’assemblage avec une résolution au niveau chromosomique et l’annotation d’un génome diploïde de Paspalum notatum. Ce résultat, issu d’un effort de collaboration entre différents partenaires argentins, italiens et français du projet MAD, constitue une étape clé pour la progression du WP3 car il servira de référence pour caractériser les génomes tétraploïdes et déterminer la structure et l’évolution de la région impliquée dans l’apomixie. Enfin, le programme de l’atelier international sur le séquençage Nanopore et la bioinformatique, organisé par le projet MAD à Rosario, en Argentine, en novembre prochain, sera discuté et décidé au cours de cette visite.
Serres végétales de l’IRD. Juan Pablo A. Ortiz (droite) & Olivier Leblanc (gauche)

Dr. Alejandra Díaz
Bahia Blanca, Argentine, CERZOS
Université de Milan (Italie), du 21 janvier 2023 au 23 mars 2023
Ce détachement avait pour objectif d’analyser l’expression de deux gènes candidats impliqués dans le mode de reproduction apomictique dans les tissus reproducteurs chez la graminée Eragrostis curvula (WP4). Pour cela, nous avons utilisé la technique d’hybridation in situ de l’ARN (RNA-ISH) pour déterminer leurs profils d’expression dans les cellules d’organes reproducteurs d’E. curvula pour un génotype apomictique et un génotype sexué. En outre, nous avons également caractérisé un gène qui montre une expression différentielle entre les plantes sexuées et apomictiques chez E. curvula et qui est également exprimé chez la plante modèle A. thaliana. Pour ce dernier objectif, nous avons aussi utilisé l’ISH pour tester l’abondance et la localisation de l’ARN de ce gène orthologue dans les tissus reproducteurs d’E. curvula (génotypes apomictique et sexué) et d’A. thaliana (génotypes diploïde et tétraploïde).
De gauche à droite: Marta Mendes, Lucia Colombo, Alejandra Díaz and Cielo Pasten
2022
Gregorio Orozco
Université de Milan, Italie
Université de Pérouse (Italie), du 2 novenbre 2022 au 2 décembre 2022
Dr. Lucia Colombo
Université de Milan, Italie
IICAR (Rosario, Argentine), du 14 novembre 2022 au 27 novembre 2022
Jimena Gallardo
Bahia Blanca, Argentine, CERZOS
Université de Pérouse (Italie), du 2 novenbre 2022 au 2 décembre 2022

Dr. Olivier Leblanc
Montpellier, France, IRD
IICAR (Argentine), du 17 octobre 2022 au 16 décembre 2022
Ce détachement vise à poursuivre (1) la caractérisation de la région chromosomique associée à l’apomixie chez les plantes fourragères tropicales. Comme nous avons maintenant terminé l’assemblage et l’annotation du génome d’un individu diploïde sexué, différentes approches seront explorées pour révéler les variations génomiques structurelles qui peuvent sous-tendre le développement apomictique chez les espèces de Paspalum. Pour celà, nous avons produit > 100 Gb de lectures longues (technologie Nanopore) à partir de plusieurs plantes de Paspalum apomictiques qui seront utilisées pour l’assemblage de novo et des analyses comparatives, et ; (2) l’analyse fonctionnelle des mutants tgs1 d’Arabidopsis et, plus particulièrement, l’analyse transcriptionnelle comparative dans les embryons d’Arabidopsis tgs1 et WT collectés à des stades précoces du développement. Ces activités font partie du WP3 et du WP4, respectivement.
Image: Clones métalliques dans la pampa proche de Rosario, Province de Santa Fe, Argentine (@ O Leblanc)

Dr. Maria Cielo Pasten
Bahia Blanca, Argentine, CERZOS
Université de Milan (Italie), du 30 septembre 2022 au 1er mars 2023
L’objectif de ce détachement était d’analyser les gènes qui montrent une expression différentielle entre ovules de plantes sexuées et apomictiques chez l’espèce Eragrostis curvula et, qui sont également présents dans la plante modèle Arabidopsis thaliana. Dans ce but, nous avons étudié le phénotype de mutants d’A. thaliana, disponibles publiquement pour certains de ces gènes, mais aussi celui de nouvelles lignées de mutants transgéniques générées par la stratégie CRISPR-Cas9. De plus, le développement des ovules dans les génotypes apomictiques et sexuels d’E. curvula a été caractérisé à l’aide de la microscopie confocale afin d’approfondir nos connaissances sur les structures et les différences entre les modes de reproduction sexuel et asexué.
De gauche à droite: Marta Mendes, Lucia Colombo, Alejandra Díaz and Cielo Pasten

Pr. Pablo Roncallo
Bahia Blanca, Argentine, CERZOS
Université de Pérouse (Italie), du 6 septembre 2022 au 8 novembre 2022
L’objectif de ce détachement est d’étendre une carte de liaison génétique tétraploïde d’Eragrostis curvula en utilisant des marqueurs DarT seq et des génotypes F1 supplémentaires afin de cartographier plus précisément la région impliquée dans l’apomixie. La population F1 a été obtenue en croisant un génotype sexuel (OTA) et un génotype apomictique (Don Walter) comme parent maternel et paternel, respectivement. Cette nouvelle carte de liaison facilitera la localisation des loci contrôlant l’apomixie (diplosporie) et permettra d’étudier l’évolution du génome de cette graminée (WP3). En collaboration avec le groupe UNIPG, j’analyse les marqueurs moléculaires (DarT seq) obtenus chez les nouveaux individus F1 et chez d’autres individus obtenus précédemment à Bahía Blanca par notre groupe pour construire une nouvelle carte de liaison intégrée, ainsi que deux cartes de liaison spécifiques aux parents. Pour cela, une analyse de ségrégation et la détection de marqueurs SNP polymorphes dans l’un ou les deux parents sont nécessaires pour résoudre ces trois cartes de liaison. De plus, la reconstruction des haplotypes en utilisant cette population F1 sera explorée ainsi que le profil SNP des parents.

Davide Perrone
Université de Milan, Italie
IICAR (Rosario, Argentine), du 2 septembre 2022 au 28 novembre 2022
Davide Perrone (Université de Milan, Italie) est accueilli au laboratoire du Prof. Silvina Pessino à IICAR CONICET, Rosario, Argentine, du 3 septembre au 27 novembre 2022. Son travail, mené en collaboration avec le Dr. Carolina Colono (IICAR-CONICET-UNR), vise à utiliser la transformation biolistique pour produire des lignées fluorescentes de Paspalum notatum DR5 (marqueur d’auxine) et TCS (marqueur de cytokinine) avec différents modes de reproduction (sexualité et apomixie aposporique). Ces matériels végétaux seront utiles pour étudier les profils hormonaux à différents stades de développement de l’ovule chez un modèle apomictique bien caractérisé. En outre, Davide contribue à l’étude du rôle du gène ARF10 (AUXIN RESPONSE FACTOR 10) dans le transport de l’auxine dans l’ovule d’Arabidopsis, en effectuant des caractérisations cyto-embryologiques de croisements arf10 x PIN1 et arf10 x DII-Venus obtenus précédemment à l’IICAR-CONICET-UNR. Enfin, il est impliqué dans l’analyse de la connexion fonctionnelle entre ARF10 et TRIMETHYL GUANOSINE SYNTHASE (TGS1), un gène impliqué dans le contrôle de l’aposporie chez Paspalum.
De gauche à droite: Carolina Colono, Silvina Pessino, Davide Perrone

Sol Vega, doctorante
Rosario, Argentine, IICAR
IRD (France), du 15 juin 2022 au 16 août 2022
Maria Sol est doctorante à l’IICAR-CONICET, Argentine, et elle visite le laboratoire du Dr. Olivier Leblanc à l’IRD (Montpellier, France). L’objectif principal de son séjour est d’initier l’analyse fonctionnelle de mutants d’Arabidopsis défectueux pour le gène IAA16, dont l’orthologue chez Paspalum notatum (PsIAA30) présente une réduction importznte de son expression pendant le développement apomictique (Siena et al., Plants 2022, 11, 472). Les mutants iaa16 hétérozygotes et homozygotes seront analysés par des observations cytoembryologiques afin de rechercher d’éventuels phénotypes reproducteurs femelles rappelant l’apomixie aposporique. Ce travail contribuera à déterminer les effets de la déplétion de IAA16 sur le développement reproductif d’Arabidopsis et à planifier une caractérisation fonctionnelle plus poussée de ce gène ou d’autres gènes appartenant à la même voie. Pendant sa visite, Maria Sol travaillera en collaboration avec le Dr Olivier Leblanc et Caroline Michaud de l’IRD, ainsi qu’avec Lorena Siena, sa directrice de thèse à l’IICAR.
Lorena Siena (gauche) & Sol Vega (droite)

Dr. Juan Pablo A. Ortiz
Rosario, Argentine, IICAR
IRD (France), du 6 juini 2022 au 7 juillet 2022
Un nouveau détachement MAD pour initier la caractérisation des génomes apomictiques (WP3) a débuté le 7 juin 2022. À cette occasion, le Dr Juan Pablo A. Ortiz de l’IICAR (CONICET-UNR, Argentine) visitera le laboratoire du Dr Olivier Leblanc à l’IRD (France). Le plan de travail comprend l’annotation d’un assemblage du génome d’un écotype diploïde de bahiagrass Pensacola (Paspalum notatum, var. saurae Parodi), obtenu précédemment dans le cadre d’une collaboration entre des laboratoires français, italiens et d’argentin. Nous avons également l’intention d’identifier et de caractériser les séquences liées au locus contrôlant l’apomixie (ACL) chez une plante tétraploïde. En particulier, nous utiliserons la technologie ONT « adaptive sampling » qui permet le séquençage de régions d’intérêt. Nous espérons donc enrichir notre séquençage avec des lectures spécifiques de la plante apomictique tétraploïde et ensuite assembler l’ACL. Olivier Leblanc, Cédric Mariac et Julie Orjuela (Unité de recherche DIADE), ainsi que Juan Manuel Vega (MAD détaché de l’IICAR) contribueront à ce travail.
Juan Pablo Ortiz (gauche) & Olivier Leblanc (droite)

Dr. Lorena Siena
Rosario, Argentine, IICAR
IRD (France), du 6 juin 2022 au 1er septembre 2022
Pendant ce détachement de 3 mois, Lorena Siena (IICAR, CONICET- Argentine) visitera l’IRD avec l’objectif de faire progresser la caractérisation fonctionnelle du gène TGS1 au cours du développement reproducteur de la plante modèle Arabidopsis thaliana. Les activités seront menées en collaboration avec le Dr. Olivier Leblanc et son groupe de l’IRD, et comprennent : des tests de complémentation utilisant la construction pTGS1:TGS1-GUS ; la détermination de l’origine des sacs embryonnaires supplémentaires observés chez les mutants tgs1 en utilisant des lignées rapportrices (pWOX2:CENH3:GFGP) à la fois de l’identité gamétophytique femelle et du nombre de chromosomes (origine réduite vs. non réduite) , et; l’analyse des données RNAseq obtenues à partir d’embryons précoce (stade globulaire) de type sauvage et mutants tgs1. Cette étude vise à déterminer les altérations des profils d’expression génique causées par la déplétion de TGS1, à identifier les gènes cibles potentiels, et à explorer si l’épissage alternatif contrôle l’embryogenèse précoce chez les plantes.
Olivier Leblanc (gauche) & Lorena Siena (droite)

Dra. Luciana Delgado
Rosario, Argentine, IICAR
IRD (France), du 20 mai 2022 au 19 août 2022
Dans le cadre de ce détachement, le Dr Luciana Delgado (IICAR) et le Dr Daphné Autran travaillent ensemble pour étudier la communication entre la division, l’expansion et la différenciation cellulaire tout au long du développement reproducteur femelle chez P. rufum, une espèce apomictique. L’objectif global est de construire un atlas 3D du développement précoce de l’ovule. Pendant le séjour, nous établirons les conditions optimales de clarification des ovules de P. rufum et les techniques de coloration de la paroi cellulaire pour obtenir des images 3D à haute résolution. Les images seront segmentées avec MorphoGraphX, classées par stade de développement, et enfin une analyse quantitative des descripteurs cellulaires sera effectuée. Nous pensons que la compréhension de la coordination entre les processus de développement reproductif avec la croissance et la division cellulaire, dans un contexte apomictique, pourrait être éclairante pour comprendre la régulation de l’apomixie.
Daphné Autran (gauche) & Luciana Delgado (droite)

Dr. Juan Pablo Selva
Bahia Blanca, Argentine, CERZOS
Université de Milan (Italie), du 16 mai 2022 au 16 juillet 2022
L’objectif de ce détachement est de poursuivre la caractérisation fonctionnelle d’un gène différentiellement exprimé dans les plantes sexuées et apomictiques d’Eragrostis curvula en utilisant des mutants de maïs portant une insertion Mutator dans le gène orthologue.
Pendant ces deux mois, j’ai collaboré avec le groupe du professeur Gabriella Consonni pour évaluer différentes populations de maïs F2 qui ont été plantées dans un champ expérimental. Les plantes ont été génotypées en utilisant des amorces spécifiques pour l’insertion du transposon située dans le premier exon du gène orthologue et des amorces spécifiques pour l’allèle de type sauvage. Les activités comprenaient également l’observation au microscope de pistils clarifiés pour rechercher des anomalies au cours du développement reproductif femelle.

Dr. Juan Pablo A. Ortiz
Rosario, Argentine, IICAR
IBBR (Italie), du 5 mai 2022 au 6 juin 2022
Un nouveau détachement MAD impliquant des chercheurs d’Argentine et d’Italie commence le 9 mai pour un mois ! En effet, le Dr Juan Pablo A. Ortiz de l’IICAR, (CONICET-UNR) est accueuilli au laboratoire du Dr Fulvio Pupilli à l‘Institut des Biosciences et des Bioressources (IBBR-CNR) de Pérugia. Le plan de travail est centré sur l’analyse fonctionnelle de ORC3, un gène qui ségrège comme complètement lié à l’apomixie, au cours de la formation de l’albumen de Paspalum simplex apomicts. En outre, il est également prévu de réaliser l’assemblage du génome de P. simplex en utilisant les long reads générés par la technologie de séquençage Oxford Nanopore. Les membres du groupe du Dr. Pupilli impliqués dans ces projets sont : María Eugenia Cáceres, Andrea Rubini, Francesca De Marchis et Michele Bellucci. A la fin du détachement, nous espérons avoir terminé la rédaction d’un manuscrit sur l’analyse fonctionnelle de PsORC3 et produit un projet d’assemblage du génome de P. simplex.
Fulvio Pupilli (gauche) & Juan Pablo A. Ortiz (droite)

Dr. Carolina Colono & Prof. Silvina Pessino
Rosario, Argentine, IICAR
Université de Milan (Italie), du 29 avril 2022 au 2 juillet 2022
Du 29 avril au 2 juillet 2022, la Dr. Carolina Colono et la Prof. Silvina Pessino visiteront le laboratoire du Prof. Lucia Colombo à l’Université de Milan, pour explorer le rôle du contrôle hormonal dans la transition de la sexualité à l’apomixie en utilisant la plante modèle Arabidopsis thaliana.
De gauche à droite: Carolina Colono, Silvina Pessino et Lucia Colombo

Juan Manuel Vega, doctorant
Rosario, Argentine, IICAR
IRD, Montpellier (France), du 19 mars 2022 au 18 juin 2022
Ce détachement s’intègre aux activités du projet MAD visant à déterminer la nature, l’évolution et le rôle fonctionnel de la région contrôlant l’aposporie (ACR) chez Paspalum notatum (voir objectifs WP3). En utilisant un génome de référence diploïde et un jeu de séquences « long reads» pour des plantes apomictiques, j’utiliserai la méthode “adaptive sampling” proposée par la plateforme ONT pour enrichir le séquençage en lectures longues de l’ACR et ensuite réaliser un assemblage à haute couverture pour une plante apomictique obligatoire et une autre apomictique facultative. Des analyses complémentaires utilisant des méthodes open source pour identifier et qualifier les variations structurelles seront également appliquées. Ces travaux devraient permettre des approches de génomique structurelle et comparative pour déterminer le contenu génique et la capacité de codage/régulation de l’ACR et, de fournir des indices sur son rôle fonctionnel dans l’émergence de l’apomixie chez Paspalum.

Dr. Diego Zappacosta
Bahia Blanca, Argentine, CERZOS
Université de Pérouse (Italie), du 1er mars 2022 au 7 mai 2022
L’objectif de ce détachement est de poursuivre l’assemblage des génomes tétraploïdes d’Eragrostis curvula pour déterminer le locus contrôlant l’apomixie et étudier l’évolution des génomes chez cette graminée (WP 3). Pendant ces deux mois, je vais analyser, en collaboration avec le groupe de l’UNIPG, les relations génétiques entre le diploïde sexué (cv Victoria), précédemment séquencé et publié par les 2 institutions, et le tétraploïde apomictique cv. Tanganyika. Nous assemblerons d’abord un pan-génome et le décomposerons en « core genome » et « dispensable genome » . Les séquences qui correspondent à la référence Victoria seront assemblées tandis que les autres seront assemblées de novo pour analyser les variations de type présence/absence. Nous analyserons ensuite les gènes orthologues pour trouver les contractions et les expansions des familles de gènes et comparerons les régions contrôlant l’apomixie chez Eragrostis et Paspalum spp. Finalement, j’appliquerai le protocole MCSeEd développé par l’UNIPG pour comparer les régions différentiellement méthylées entre les génotypes apomictiques et sexuels (Task 3.3).
